leetcode-27-exercise_bit maniputation
461. Hamming Distance

解题思路:
把两个数的每一位和1比较,如果结果不同说明这两位不同。要比较32次。
int hammingDistance(int x, int y) {
int result = 0;
for (int i = 0; i < 32; i++) {
if (((x>>i)&0x1) != ((y>>i)&0x1)) {
result ++;
}
}
return result;
}
477. Total Hamming Distance
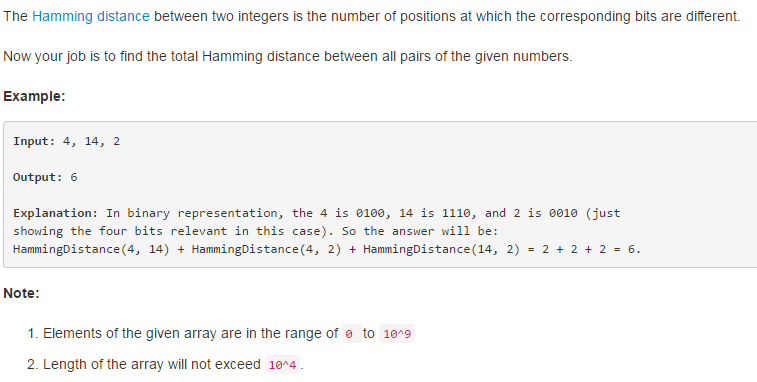
解题思路:
因为数据是从0到10^9的,所以可以转化为31位二进制数(10^9 = (10^3)^3 ~ (2^10)^3 = 2^30)。对于所有数的每一位,
计算该位置上1的个数和0的个数,那么,这一位的总差异数应该是二者之积。取每一位的话,可以用右移来取。
int totalHammingDistance(vector<int>& nums) {
int result = 0;
int ones = 0;
for (int i = 0; i < 31; i++) {
for (int j = 0; j < nums.size(); j++) {
if ((nums[j] >> i) & 0x1)
ones ++;
}
result += ones * (nums.size() - ones);
ones = 0;
}
return result;
}
78. Subsets
Given a set of distinct integers, nums, return all possible subsets.
Note: The solution set must not contain duplicate subsets.
解题思路:
首先,子集的数量应该是2^n。注意创建这种结构的写法。
对于result来说,nums的每一个元素都可能存在也可能不存在。如果把j写成二进制,如果第i位为1,就可以把nums[i]
放入result[j]。
vector<vector<int>> subsets(vector<int>& nums) {
int size = pow(2, nums.size());
vector<vector<int> > result(size, vector<int>{});
for (int i = 0; i < nums.size(); i++) {
for (int j = 0; j < size; j++) {
if (j >> i & 0x1)
result[j].push_back(nums[i]);
}
}
return result;
}
201. Bitwise AND of Numbers Range

解题思路:
要想确定整个范围内的数,转换为二进制时各个位置是否全为1,全部写出来是没有必要的。注意,此处只需要找出起始数共同的前缀就好了,
因为两个数可以修改后面的部分,必然会存在有0的位置,所以通过右移找出共同前缀,记录右移次数,再左移回来就好。
int rangeBitwiseAnd(int m, int n) {
int i = 0;
while (m != n) {
m = m >> 1;
n = n >> 1;
i ++;
}
return (m << i);
}
187. Repeated DNA Sequences

解题思路:
使用unordered_map。其中size_t是一个与机器相关的unsigned类型,其大小足以保证存储内存中对象的大小。
用hash存子串,节省查找时间。如果子串重复次数等于2,就保留。如果这个子串重复次数多于2了,那肯定保存过了,就不用管了。
vector<string> findRepeatedDnaSequences(string s) {
vector<string> result;
if (s.length() <= 10)
return result;
hash<string> h;
unordered_map<size_t, int> m;
for (int i = 0; i <= s.length() - 10; i++) {
string sub = s.substr(i, 10);
m[h(sub)] ++;
if (m[h(sub)] == 2) {
result.push_back(sub);
continue;
}
}
return result;
}

