转:谱聚类(spectral clustering)及其实现详解
转自:https://blog.csdn.net/yc_1993/article/details/52997074
Preface
开了很多题,手稿都是写好一直思考如何放到CSDN上来,一方面由于公司技术隐私,一方面由于面向对象不同,要大改,所以一直没贴出完整,希望日后可以把开的题都补充全。
先把大纲列出来:
一、从狄多公主圈地传说说起
二、谱聚类的演算
(一)、演算
1、谱聚类的概览
2、谱聚类构图
3、谱聚类切图
(1)、RatioCut
(2)、Ncut
(3)、一点题外话
(二)、pseudo-code
三、谱聚类的实现(scala)
(一)、Similarity Matrix
(二)、kNN/mutual kNN
(三)、Laplacian Matrix
(四)、Normalized
(五)、Eigenvector(Jacobi methond)
(六)、kmeans/GMM
四、一些参考文献
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
一、从狄多公主圈地传说说起
谱聚类(spectral clustering)的思想最早可以追溯到一个古老的希腊传说,话说当时有一个公主,由于其父王去世后,长兄上位,想独揽大权,便杀害了她的丈夫,而为逃命,公主来到了一个部落,想与当地的酋长买一块地,于是将身上的金银财宝与酋长换了一块牛皮,且与酋长约定只要这块牛皮所占之地即可。聪明的酋长觉得这买卖可行,于是乎便答应了。殊不知,公主把牛皮撕成一条条,沿着海岸线,足足围出了一个城市。
故事到这里就结束了,但是我们要说的才刚刚开始,狄多公主圈地传说,是目前知道的最早涉及Isoperimetric problem(等周长问题)的,具体为如何在给定长度的线条下围出一个最大的面积,也可理解为,在给定面积下如何使用更短的线条,而这,也正是谱图聚类想法的端倪,如何在给定一张图,拿出“更短”的边来将其“更好”地切分。而这个“更短”的边,正是对应了spectral clustering中的极小化问题,“更好”地切分,则是对应了spectral clustering中的簇聚类效果。
谱聚类最早于1973年被提出,当时Donath 和 Hoffman第一次提出利用特征向量来解决谱聚类中的f向量选取问题,而同年,Fieder发现利用倒数第二小的特征向量,显然更加符合f向量的选取,同比之下,Fieder当时发表的东西更受大家认可,因为其很好地解决了谱聚类极小化问题里的NP-hard问题,这是不可估量的成就,虽然后来有研究发现,这种方法带来的误差,也是无法估量的,下图是Fielder老爷子,于去年15年离世,缅怀。
二、谱聚类的演算
(一)、演算
1、谱聚类概览
谱聚类演化于图论,后由于其表现出优秀的性能被广泛应用于聚类中,对比其他无监督聚类(如kmeans),spectral clustering的优点主要有以下:
1.过程对数据结构并没有太多的假设要求,如kmeans则要求数据为凸集。
2.可以通过构造稀疏similarity graph,使得对于更大的数据集表现出明显优于其他算法的计算速度。
3.由于spectral clustering是对图切割处理,不会存在像kmesns聚类时将离散的小簇聚合在一起的情况。
4.无需像GMM一样对数据的概率分布做假设。
- 1
- 2
- 3
- 4
- 5
同样,spectral clustering也有自己的缺点,主要存在于构图步骤,有如下:
1.对于选择不同的similarity graph比较敏感(如 epsilon-neighborhood, k-nearest neighborhood,fully connected等)。
2.对于参数的选择也比较敏感(如 epsilon-neighborhood的epsilon,k-nearest neighborhood的k,fully connected的 )。
- 1
- 2
- 3
谱聚类过程主要有两步,第一步是构图,将采样点数据构造成一张网图,表示为G(V,E),V表示图中的点,E表示点与点之间的边,如下图:
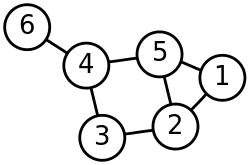
图1 谱聚类构图(来源wiki)
第二步是切图,即将第一步构造出来的按照一定的切边准则,切分成不同的图,而不同的子图,即我们对应的聚类结果,举例如下:
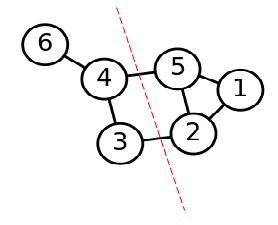
图2 谱聚类切图
初看似乎并不难,但是…,下面详细说明推导。
2、谱聚类构图
在构图中,一般有三种构图方式:
1. εε-neighborhood
2. k-nearest neighborhood
3. fully connected
前两种可以构造出稀疏矩阵,适合大样本的项目,第三种则相反,在大样本中其迭代速度会受到影响制约,在讲解三种构图方式前,需要引入similarity function,即计算两个样本点的距离,一般用欧氏距离:si,j=∥xi−xj∥2si,j=‖xi−xj‖2, si,jsi,j表示样本点xixi与xjxj的距离,或者使用高斯距离si,j=e−∥∥xi−xj∥∥22σ2si,j=e−‖xi−xj‖22σ2,其中σσ 的选取也是对结果有一定影响,其表示为数据分布的分散程度,通过上述两种方式之一即可初步构造矩阵S:Si,j=[s]i,jS:Si,j=[s]i,j,一般称 为Similarity matrix(相似矩阵)。
对于第一种构图εε -neighborhood,顾名思义是取si,j≤εsi,j≤ε的点,则相似矩阵SS可以进一步重构为邻接矩阵(adjacency matrix)WW :
可以看出,在εε-neighborhood重构下,样本点之间的权重没有包含更多的信息了。
对于第二种构图k-nearest neighborhood,其利用KNN算法,遍历所有的样本点,取每个样本最近的k个点作为近邻,但是这种方法会造成重构之后的邻接矩阵WW非对称,为克服这种问题,一般采取下面两种方法之一:
一是只要点xixi在xjxj的K个近邻中或者xjxj在xixi的K个近邻中,则保留si,jsi,j,并对其做进一步处理WW,此时 为:
二是必须满足点 在 的K个近邻中且 在 的K个近邻中,才会保留si,jsi,j并做进一步变换,此时WW为:
对于第三种构图fully connected,一般使用高斯距离: si,j=e−∥∥xi−xj∥∥22σ2si,j=e−‖xi−xj‖22σ2,则重构之后的矩阵WW与之前的相似矩阵SS相同,为:Wi,j=Si,j=[s]i,jWi,j=Si,j=[s]i,j。
在了解三种构图方式后,还需要注意一些细节,对于第一二中构图,一般是重构基于欧氏距离的 ,而第三种构图方式,则是基于高斯距离的 ,注意到高斯距离的计算蕴含了这样一个情况:对于∥xi−xj∥2‖xi−xj‖2比较大的样本点,其得到的高斯距离反而值是比较小的,而这也正是SS可以直接作为WW的原因,主要是为了将距离近的点的边赋予高权重。
得到邻接矩阵WW后,需要做进一步的处理:
(1).计算阶矩(degree matrix)DD:
其中其中wi,jwi,j为邻接矩阵WW元素,∑jwi,j∑jwi,j表示将图中某点所连接的其他点的边的权重之和,可以看出,DD为对角矩阵.
(2).计算拉普拉斯矩阵(Laplacians matrix)LL:L=D−WL=D−W
如此,在构图阶段基本就完成了,至于为什么要计算出拉普拉斯矩阵LL,可以说L=D−WL=D−W这种形式对于后面极大化问题是非常有利的(不知道前人是怎么想到的,不然LL也不会被命名为拉普拉斯了吧)。
3、谱聚类切图
谱聚类切图存在两种主流的方式:RatioCut和Ncut,目的是找到一条权重最小,又能平衡切出子图大小的边,下面详细说明这两种切法。
在讲解RatioCut和Ncut之前,有必要说明一下问题背景和一些概念,假设V为所有样本点的集合,{A1,A2,⋯,Ak}{A1,A2,⋯,Ak}表示V的子集集合,其中A1∪A2∪⋯∪Ak=V且A1∩A2∩⋯∩Ak=∅A1∪A2∪⋯∪Ak=V且A1∩A2∩⋯∩Ak=∅,则子集与子集之间连边的权重和为:
其中Ai¯Ai¯为AiAi的补集,意为除AiAi子集外其他V的子集的并集,W(Ai,Ai¯)W(Ai,Ai¯)为AiAi与其他子集的连边的和,为:
其中wm,nwm,n为邻接矩阵W中的元素。
由于我们切图的目的是使得每个子图内部结构相似,这个相似表现为连边的权重平均都较大,且互相连接,而每个子图间则尽量没有边相连,或者连边的权重很低,那么我们的目的可以表述为:
但是稍微留意可以发现,这种极小化的切图存在问题,如下图:
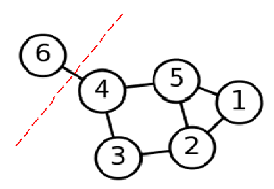
图3 问题切图
如上图,如果用mincut(A1,A2,⋯,Ak)mincut(A1,A2,⋯,Ak)这种方法切图,会造成将V切成很多个单点离散的图,显然这样是最快且最能满足那个最小化操作的,但这明显不是想要的结果,所以有了RatioCut和Ncut,具体地:
其中|Ai||Ai| 为AiAi中点的个数,vol(Ai)vol(Ai)为AiAi中所有边的权重和。
下面详细讲解这两种方法的计算:Ratiocut & Ncut
(1).Ratiocut
Ratiocut切图考虑了目标子图的大小,避免了单个样本点作为一个簇的情况发生,平衡了各个子图的大小。Ratiocut的目标同样是极小化各子图连边和,如下:
对上述极小化问题做一下转化,引入{A1,A2,⋯,Ak}{A1,A2,⋯,Ak}的指示向量hj={h1,h2,⋯,hi,⋯,hk},j=1,2,⋯,khj={h1,h2,⋯,hi,⋯,hk},j=1,2,⋯,k. 其中i表示样本下标,j表示子集下标, 表示样本i对子集j的指示,具体为:
通俗理解就是,每个子集AjAj对应一个指示向量hjhj,而每个hjhj里有N个元素,分别代表N个样本点的指示结果,如果在原始数据中第i个样本被分割到子集AjAj里,则hjhj的第i个元素为1|Aj|√1|Aj|,否则为0。
进一步,计算 ,可以得到:


