论文《Learning Properties of Ordered and Disordered Materials from Multi-fidelity Data》中的代码实现
github地址:https://github.com/materialsvirtuallab/megnet/tree/master/multifidelity#issues
介绍:当前的存储库利用了由同一作者开发的现有MEGNET软件包,并将MEGNET功能扩展到多保真数据集的建模。该存储库将共享公开发布的多保真带隙数据,并展示了运行多保真数据集的模型拟合的示例。
需要的包或库:
pymatgen>=2020.7.18
pandas>=1.0.5
tensorflow-gpu>=2.0.0
numpy>=1.19.1
monty>=3.0.4
megnet>=1.1.8
问:conda的创建的虚拟环境 和 venv创建的虚拟环境 有什么区别?
答:
包管理工具:
- Conda本身是一个包管理工具,能创建和管理虚拟环境。
- venv创建的虚拟环境使用Python的标准包管理系统pip。
Python解释器:
- Conda随环境下载和安装指定版本的Python解释器。
- venv复制现有的Python解释器。
平台无关性:
- Conda是跨平台的包管理系统。
- venv是Python的标准库模块,平台无关。
生态系统:
- Conda适用于跨平台、语言和复杂依赖关系的管理。
- venv通常更轻量级,适用于纯Python项目。
因为本linux系统中已经存在了python3.11的解释器,但是该项目需要用到python3.7.9,因此使用Conda创建虚拟环境可以再虚拟环境中指定python版本,并且conda可以管理复杂依赖关系,因此这里使用conda更加合适。
一、配置环境
1.确保已安装 Anaconda 或 Miniconda

2.创建虚拟环境
使用 conda create 命令来创建一个新的虚拟环境,并指定 Python 版本为 3.7.9 给环境命名为 myenv
conda create --name myenv python=3.7.9
3.激活虚拟环境
创建完环境后,激活它:
conda activate myenv
4.检查当前的 Python 和 pip 路径,以确认它们指向虚拟环境
(myenv) ubuntu@ubuntu-System-Product-Name:~/zdn$ which python
/home/ubuntu/anaconda3/envs/myenv/bin/python
(myenv) ubuntu@ubuntu-System-Product-Name:~/zdn$ which pip
/home/ubuntu/.local/bin/pip
结果可见: pip 指向的是 /home/ubuntu/.local/bin/pip,而不是虚拟环境中的 pip。这表明 pip 可能没有正确安装到虚拟环境中
解决:
重新安装pip conda install pip 如果已经安装,可以使用以下命令来强制重新安装 conda install --force-reinstall pip
接着检查pip路径 which pip还是不行,
/home/ubuntu/.local/bin/pip 是旧版本的 pip,可以尝试删除它,确保不会影响到虚拟环境的使用。使用以下命令删除 rm -f /home/ubuntu/.local/bin/pip
然后再重复上述步骤(安装或重新安装 pip)以确保在虚拟环境中拥有一个干净的 pip 安装

还可以尝试直接通过 Python 模块执行 pip,以确保使用的是虚拟环境中的 pip python -m pip --version

5.安装依赖
pip install -r requirements-gpu.txt
注意:在 conda 虚拟环境中,使用 pip 来安装依赖
结果网络不可达,因此使用国内的镜像网站下载:
pip install -i https://pypi.tuna.tsinghua.edu.cn/simple -r requirements-gpu.txt
结果提示:
The "tensorflow-gpu" package has been removed!
Please install "tensorflow" instead.
Other than the name, the two packages have been identical
since TensorFlow 2.1, or roughly since Sep 2019. For more
information, see: pypi.org/project/tensorflow-gpu
修改requirements-gpu.txt文件内容:把tensorflow-gpu改成tensorflow:
pymatgen>=2020.7.18
pandas>=1.0.5
tensorflow>=2.0.0
numpy>=1.19.1
monty>=3.0.4
megnet>=1.1.8
再重新执行pip install -i https://pypi.tuna.tsinghua.edu.cn/simple -r requirements-gpu.txt即可
6.验证安装
验证 Python 版本和已安装的库是否正确:
python --version 应该显示 Python 3.7.9
pip list 查看已安装的库
- 如果要退出虚拟环境,可以使用以下命令:
conda deactivate
- 如果要删除虚拟环境,可以使用以下命令:
conda remove --name myenv --all
二、数据集字段解释
论文中使用的完整数据在data_no_structs.json.gz中
提示:在vscode中,把光标置于其中一个花括号或其他符号上,使用 Ctrl + Shift + \(Windows/Linux)或 Cmd + Shift + \(macOS),使得光标跳转到对应的符号上。
1.qm7b.json
qm7b.json(这里提取了根节点"molecules"和"targets"以及它们的其中几条数据;molecules:“1849”和“4855”等;targets:1-7211)
{"molecules": {
"1849": {"@module": "pymatgen.core.structure", "@class": "Molecule", "charge": 0.0, "spin_multiplicity": 1, "sites": [{"name": "C", "species": [{"element": "C", "occu": 1}], "xyz": [2.571301, -0.368033, -0.031506], "properties": {}}, {"name": "C", "species": [{"element": "C", "occu": 1}], "xyz": [1.117728, -0.021742, -0.013405], "properties": {}}, {"name": "C", "species": [{"element": "C", "occu": 1}], "xyz": [0.559635, 1.197419, -0.010524], "properties": {}}, {"name": "N", "species": [{"element": "N", "occu": 1}], "xyz": [-0.847177, 1.131975, 0.079215], "properties": {}}, {"name": "C", "species": [{"element": "C", "occu": 1}], "xyz": [-1.277761, -0.196208, 0.013988], "properties": {}}, {"name": "N", "species": [{"element": "N", "occu": 1}], "xyz": [-2.460674, -0.671719, -0.060165], "properties": {}}, {"name": "C", "species": [{"element": "C", "occu": 1}], "xyz": [-0.00348, -1.040709, 0.050603], "properties": {}}, {"name": "H", "species": [{"element": "H", "occu": 1}], "xyz": [3.194635, 0.531377, -0.082546], "properties": {}}, {"name": "H", "species": [{"element": "H", "occu": 1}], "xyz": [2.825737, -1.001159, -0.893161], "properties": {}}, {"name": "H", "species": [{"element": "H", "occu": 1}], "xyz": [2.86786, -0.928585, 0.866575], "properties": {}}, {"name": "H", "species": [{"element": "H", "occu": 1}], "xyz": [1.044647, 2.166435, -0.029022], "properties": {}}, {"name": "H", "species": [{"element": "H", "occu": 1}], "xyz": [-1.449759, 1.902035, -0.173852], "properties": {}}, {"name": "H", "species": [{"element": "H", "occu": 1}], "xyz": [-3.161008, 0.077357, -0.067436], "properties": {}}, {"name": "H", "species": [{"element": "H", "occu": 1}], "xyz": [0.004031, -1.75587, -0.781669], "properties": {}}, {"name": "H", "species": [{"element": "H", "occu": 1}], "xyz": [0.024283, -1.637735, 0.97283], "properties": {}}]},
"4855": {...}
...
"765":{...}
}
"targets": {
"1": {"HF": {"sto3g": 19.57, "631g": 4.92, "ccpvdz": 6.35}, "MP2": {"sto3g": 4.62, "631g": 2.15, "ccpvdz": 8.55}, "CCSD(T)": {"sto3g": 6.66, "631g": 3.25, "ccpvdz": 8.38}},
"2": {"HF": {"sto3g": 12.81, "631g": 2.03, "ccpvdz": 2.81}, "MP2": {"sto3g": -1.18, "631g": 0.02, "ccpvdz": 5.76}, "CCSD(T)": {"sto3g": 1.01, "631g": 1.24, "ccpvdz": 5.43}},
"3": {"HF": {"sto3g": 39.97, "631g": 12.94, "ccpvdz": 13.24}, "MP2": {"sto3g": 28.92, "631g": 13.93, "ccpvdz": 19.26}, "CCSD(T)": {"sto3g": 25.84, "631g": 11.83, "ccpvdz": 16.62}},
"4": {...}
...
"7211":{...}
}
}
以下是"molecules"中的第一条数据“1849”的可读性更强的形式:
"1849": {
"@module": "pymatgen.core.structure",
"@class": "Molecule",
"charge": 0.0,
"spin_multiplicity": 1,
"sites": [
{
"name": "C",
"species": [
{
"element": "C",
"occu": 1
}
],
"xyz": [2.571301, -0.368033, -0.031506],
"properties": {}
},
{
"name": "C",
"species": [
{
"element": "C",
"occu": 1
}
],
"xyz": [1.117728, -0.021742, -0.013405],
"properties": {}
},
{
"name": "C",
"species": [
{
"element": "C",
"occu": 1
}
],
"xyz": [0.559635, 1.197419, -0.010524],
"properties": {}
},
{
"name": "N",
"species": [
{
"element": "N",
"occu": 1
}
],
"xyz": [-0.847177, 1.131975, 0.079215],
"properties": {}
},
{
"name": "C",
"species": [
{
"element": "C",
"occu": 1
}
],
"xyz": [-1.277761, -0.196208, 0.013988],
"properties": {}
},
{
"name": "N",
"species": [
{
"element": "N",
"occu": 1
}
],
"xyz": [-2.460674, -0.671719, -0.060165],
"properties": {}
},
{
"name": "C",
"species": [
{
"element": "C",
"occu": 1
}
],
"xyz": [-0.00348, -1.040709, 0.050603],
"properties": {}
},
{
"name": "H",
"species": [
{
"element": "H",
"occu": 1
}
],
"xyz": [3.194635, 0.531377, -0.082546],
"properties": {}
},
{
"name": "H",
"species": [
{
"element": "H",
"occu": 1
}
],
"xyz": [2.825737, -1.001159, -0.893161],
"properties": {}
},
{
"name": "H",
"species": [
{
"element": "H",
"occu": 1
}
],
"xyz": [2.86786, -0.928585, 0.866575],
"properties": {}
},
{
"name": "H",
"species": [
{
"element": "H",
"occu": 1
}
],
"xyz": [1.044647, 2.166435, -0.029022],
"properties": {}
},
{
"name": "H",
"species": [
{
"element": "H",
"occu": 1
}
],
"xyz": [-1.449759, 1.902035, -0.173852],
"properties": {}
},
{
"name": "H",
"species": [
{
"element": "H",
"occu": 1
}
],
"xyz": [-3.161008, 0.077357, -0.067436],
"properties": {}
},
{
"name": "H",
"species": [
{
"element": "H",
"occu": 1
}
],
"xyz": [0.004031, -1.75587, -0.781669],
"properties": {}
},
{
"name": "H",
"species": [
{
"element": "H",
"occu": 1
}
],
"xyz": [0.024283, -1.637735, 0.97283],
"properties": {}
}
]
}
解释:
这段JSON数据是关于分子molecules的结构信息,具体内容涉及到该分子的原子组成、坐标等信息。下面是对主要组件的解释:
"molecules": 这是根节点,分子的信息集合。
"1849": 某个分子式的唯一标识符(ID)。
"@module": "pymatgen.core.structure": 指明了该数据使用的模块,pymatgen 是一个用于材料科学的 Python 库。
"@class": "Molecule": 表示该数据作为一个 Molecule (分子)类实例。
"charge": 0.0: 表示该分子是中性的,即没有净电荷。
"spin_multiplicity": 1: 表示该分子的自旋多重性为1,意味着它是单态,通常意味着所有电子都是配对的。
"sites":数组,该分子中各个原子的位置信息、详细信息;
每个原子又通过一个对象表示,包含以下字段:
"name": 原子的名称,如 "C"(碳)、"N"(氮)、"H"(氢)。
"species":是一个数组,表示与一个特定原子相关的物种信息。在上述所给例子中,数组中均只有一个对象
"element": "C":表示该原子的元素符号为 "C",即碳(Carbon)。这表明当前位置上的原子是碳原子。
"occu": 1:表示该位置上有一个碳原子。一般来说,"occu"(occupancy)用来指示某个位置被特定类型的原子占据的程度或数量。这里值为1意味着该位置上只存在一个碳原子。
"element": 原子的元素符号。
"occu": 占据情况,值为1表示该位置上有一个原子。
"xyz":三维坐标,表示原子在空间中的位置。例如:"xyz": [2.571301, -0.368033, -0.031506]这表示第一个碳原子的三维坐标,分别为 x, y, z 轴的值。
"properties":这里是一个空对象 {},可以在需要时添加与原子相关的其他属性信息。
要确定"1849" 这个分子的化学式,需要从 "sites" 部分提取所有原子的数量和种类。
在提供的数据中,包含的元素有:
碳 (C)
氮 (N)
氢 (H)
原子计数:
碳 (C): 6 个
氮 (N): 2 个
氢 (H): 8 个
将这些信息结合起来,可以得到该分子的化学式:C6H8N2
以下是targets的第一条数据:
"1": {"HF": {"sto3g": 19.57, "631g": 4.92, "ccpvdz": 6.35}, "MP2": {"sto3g": 4.62, "631g": 2.15, "ccpvdz": 8.55}, "CCSD(T)": {"sto3g": 6.66, "631g": 3.25, "ccpvdz": 8.38}},
解释:
"targets":这是一个包含多个目标的字典(或对象)。
HF:指的是 Hartree-Fock 方法,这是一种量子化学计算方法,用于估计电子结构。
sto3g、631g、ccpvdz:这些是不同的基组(一套数学工具),即用于计算化学中用来描述分子中电子行为的一套工具。每个基组有其特定的特点:
sto3g:计算速度很快,但可能不够精确,适合快速初步估算或者小分子的计算。
631g:计算较为复杂,可以提供更精确的结果,比如中等大小的分子。
ccpvdz:适合于需要极高精确度的计算,像大型分子或重要的反应机制。
每个基组下的数值(如 19.57, 4.92, 6.35)表示使用该方法和基组得到的能量或其他相关物理量(例如总能量、能量差等)。
MP2:一种改进的量子化学方法,通常在 HF 的基础上进一步修正,以提高计算精度。
CCSD(T):指的是耦合簇理论的单重态/双重态/三重态 (Coupled Cluster with Single and Double excitations, plus perturbative Triple excitations)。这是非常精确的量子化学方法之一,通常用于处理多电子系统。
各个字段的示例,以目标 "1" 为例:
"1": {
"HF": {
"sto3g": 19.57,
"631g": 4.92,
"ccpvdz": 6.35
},
"MP2": {
"sto3g": 4.62,
"631g": 2.15,
"ccpvdz": 8.55
},
"CCSD(T)": {
"sto3g": 6.66,
"631g": 3.25,
"ccpvdz": 8.38
}
}
在 HF 方法中,基组 sto3g 得到的值是 19.57,631g 是 4.92,而 ccpvdz 是 6.35,MP2 和 CCSD(T) 同理。
这个数据集是比较不同计算方法在不同基组下对某些材料或分子的计算结果的性能。
2.G4MP2.json
"G4MP2": {
"U0": {"4841": -67.01220863280132, "4556": -67.21779075980045, "5128": -67.87361271120126, "3018": -67.09144822960079, "5807": -69.023049458601, "1414":... , ... , "3405": -67.49684366680124},
"molecules": {
"4841": {
"@module": "pymatgen.core.structure",
"@class": "Molecule", "charge": 0.0,
"spin_multiplicity": 1,
"sites": [
{"name": "C",
"species": [{"element": "C", "occu": 1}],
"xyz": [-1.4340938197, -0.0415774452, -2.2842168124],
"properties": {}},
{"name": "C",
"species": [{"element": "C", "occu": 1}],
"xyz": [-1.885835766, -0.0053961018, -0.8235225225],
"properties": {}},
{...}
...
{"name": "H",
"species": [{"element": "H", "occu": 1}],
"xyz": [-1.0738297846, -2.0310535944, 0.2235490316],
"properties": {}}
]
},
"4556": {...},
...
"3405":{...}
}
"B3LYP": {
"U0":{
"89837": -76.04296479640053,
"119346": -86.89253093600009,
"80768": -79.70700701780105,
"97825": -82.31560066740121,
"92513": -84.45648477380048,
"42229": -66.80918437739882,
"98745": -68.80358951759978,
"113078": -91.39892925580071,
...
"94726": -78.01173679780123
},
"molecules":{
"89837": {
"@module": "pymatgen.core.structure",
"@class": "Molecule",
"charge": 0.0,
"spin_multiplicity": 1,
"sites": [
{
"name": "O",
"element": "O",
"occu": 1,
"xyz": [0.3518, 1.0551, 0.5891]
},
{
"name": "C",
"element": "C",
"occu": 1,
"xyz": [0.0055, -0.1833, 0.0355]
},
...
{
"name": "H",
"element": "H",
"occu": 1,
"xyz": [1.9817, -0.1119, -2.6350]
}
]
},
"119346": {},
"80768": {},
...
"94726":{}
}
解释:
"G4MP2" 和 "B3LYP":这两个字段表示不同的量子化学计算方法。G4MP2 是一种基于多体微扰理论的高精度方法,而 B3LYP 是一种广泛使用的混合密度泛函理论(DFT)方法。
"U0":包含多个键值对,其中每个键(如 "4841", "4556" 等)通常代表特定的分子唯一标识符;每个值为相应分子的零点能量(通常以 kcal/mol 或 eV 为单位),是该方法下的计算结果。
"molecules":与每种计算相关联的分子结构信息,类似于数据库中的记录。每个键(如 "4841", "4556" 等)对应一个分子的唯一标识符,与 U0 中的标识符一致。
具体的分子数据结构
"@module":指定了该对象的模块路径,表明该数据结构属于 pymatgen 库(Python Materials Genomics)。
"@class":表示该对象的类名,这里是 "Molecule",表明这是一个分子对象。
"charge":分子的电荷数。0.0 表示这是一个中性分子。
"spin_multiplicity":自旋重数,指分子的自旋状态。例如,1 表示该分子是单重态(没有未配对电子)。
"sites":这个字段包含一个数组,其中每个元素表示分子中的一个原子或原子团,包括以下信息:
"name": 原子的名称(例如 "C" 表示碳,"H" 表示氢等)。
"species": 包含与原子相关的其他数据,通常包括元素类型和占据情况(occu)。
"element": 原子的化学元素符号。
"occu": 原子的占据状态(通常为1表示完整占据)。
"xyz": 原子的三维坐标,以列表形式给出(x, y, z坐标)。
"properties": 可用于存储额外的原子属性或信息,但此例中为空。
这个 JSON 数据集主要用于存储基于不同计算方法的分子结构及其能量信息。
3.data_no_structures.json
{
"pbe": {
"mp-647999": 1.7049999999999996,
"mp-764266": 0.8498,
"mp-19019": 2.1744000000000003,
"mp-685999": 0.0,
"mp-767290": 1.8454999999999995,
"mp-11919": 1.8494000000000002,
"mp-773604": 1.0233999999999999,
...
"mp-973994": 0.0
},
"hse": {
"mp-19019": 2.612554,
"mp-776233": 2.0233980000000003,
"mp-11919": 2.960902,
"mp-2589": 6.04121,
"mp-28153": 3.477344,
"mp-322": 1.356125,
...
"mp-771096": 2.478525
},
"gllb-sc": {
"mp-17918": 4.22,
"mp-505702": 5.83,
"mp-18373": 4.85,
"mp-13982": 1.51,
"mp-10919": 2.95,
"mp-11919": 4.03,
"mp-28883": 6.47,
...
"mp-27394": 5.01
},
"scan": {
"mp-557056": 1.5574,
"mp-9900": 0.3162,
"mp-19318": 2.6264,
"mp-353": 0.2562,
"mp-5495": 0.3055,
"mp-610517": 0.8849,
...
"mp-2076": 0.7161
},
"ordered_exp": {
"icsd-44914": 1.3,
"icsd-629000": 0.355,
"icsd-670493": 3.4028090909090905,
"icsd-196824": 0.11744444444444443,
"icsd-81658": 2.26,
"icsd-420557": 1.8,
"icsd-41445": 0.22063157894736837,
"icsd-79025": 8.0,
...
"icsd-159134": 0.0
},
"disordered_exp": {
"icsd-24770": 2.998325,
"icsd-78345": 2.753,
"icsd-431153": 2.18,
"icsd-4315": 4.77,
"icsd-73255": 3.255557142857142,
"icsd-80635": 2.7999999999999994,
...
"icsd-23379": 0.0
}
}
解释:
"pbe":
"mp-647999": 1.7049999999999996
mp_id: 这是每种材料在 Materials Project 数据库中的唯一标识码
1.7049999999999996:pbe方法计算得到的带隙值
"hse" : 同理,hse方法得到的带隙值
"gllb-sc":同理
"scan":同理
"ordered_exp":有序结构的实验数据
"icsd-44914": 1.3
icsd_id: 这是每种材料在 ICSD 数据库中的唯一标识码
1.3:实验测量得到的带隙值
"disordered_exp":无序结构的实验数据
icsd_id:Exp band gap 同理
-
在这个数据集中,mp_id是来自 Materials Project 的材料 ID。例如,氯化钠(NaCl)的 mp_id 是 mp-22862;此外,还有另一个标识符是 icsd_id,它来源于国际晶体结构数据库(ICSD),该数据库的信息均为实验测量得到的数据。
-
data['pbe']['mp-22862'] 可以用来获取 NaCl 的 PBE 方法计算出的带隙值。在这个例子中,NaCl 的 PBE 带隙是 5.003 eV
-
在data_no_structures.json中没有结构信息。
- 如果想获取与特定 mp_id 对应的材料结构,可以使用 pymatgen 提供的 REST API。这个工具可以帮你从 Materials Project 获取有关该材料的详细信息和结构数据;
- 而ICSD 结构的数据需要商业访问权限,作者的github中未给出,需要自己获取。
4.各种方法比较
问:pbe、hse、gllb-sc、scan与 Hartree-Fock 、MP2、CCSD(T)方法以及G4MP2、B3LYP、qm7b方法有什么关系
答:
这些方法都是计算化学和材料科学中用于预测分子或材料性质的计算方法,但它们各自基于不同的理论框架和近似。下面是对这些方法的简要解释以及它们之间的关系:
(1)DFT 方法
- PBE (Perdew-Burke-Ernzerhof):
PBE 是一种广泛使用的密度泛函理论(DFT)的方法,属于广义梯度近似(GGA)。DFT 方法通过电子密度来描述系统,相比传统波函数方法,它通常计算更快,但在某些情况下精度较低,尤其是在描述范德瓦尔斯力和激发态等方面。
- HSE (Heyd-Scuseria-Ernzerhof):
HSE 是一种混合密度泛函方法,它结合了局部密度(或者 GGA)与 Hartree-Fock 理论的部分贡献。这种方法通常可以提供更准确的带隙计算,特别适用于半导体和绝缘体的研究。
- GLLB-SC (Gritsenko-Lindh-Bultinck-Baerends Self-Consistent):
GLLB-SC 是一种改进的 DFT 方法,其目标是提高能量和带隙计算的准确性。它通过自洽地考虑电荷转移等效应来改善计算结果。
- SCAN (Strongly Constrained and Appropriately Normed):
SCAN 是一种新型的 DFT 方法,旨在克服传统 GGA 方法的一些局限,通过强约束条件和适当规范,更好地描述非均匀电子气体的特性。
(2)波函数方法
- HF(Hartree-Fock):
一种基础的量子化学方法,通过自洽场理论来近似多电子系统的波函数。虽然 Hartree-Fock 方法简单且计算快速,但它不能很好地处理电子关联,特别是在强关联体系中。
- MP2 (Møller-Plesset Perturbation Theory, 2nd order):
MP2 是一种基于 Hartree-Fock 的微扰理论方法,可用于考虑电子关联的影响,比 Hartree-Fock 更加精确,但计算成本也显著增加。
- CCSD(T) (Coupled Cluster with Single and Double excitations and perturbative Triple corrections):
CCSD(T) 是一种高精度的量子化学方法,能够非常准确地考虑电子关联,因此在量子化学中被广泛认为是“金标准”之一。
(3)其他方法
- G4MP2:
G4MP2 是一种基于多层次方法的组合模型,旨在提供高精度的热力学数据和反应路径的估计。它是对经典 MP2 方法的改进。
- B3LYP:
B3LYP 是一种混合密度泛函方法,结合了 Hartree-Fock 和 DFT 中的局部密度近似。它在许多化学领域被广泛使用。
- QM7b:
QM7b 是一个用于机器学习的分子数据集,包含了许多小分子的计算化学数据,通常用于机器学习模型的训练和验证。
- 实验测量方法
(4)关系总结
-
密度泛函理论(DFT)(如 PBE、HSE、GLLB-SC 和 SCAN)通常用于计算材料和分子的电子结构,具有较快的计算速度,但在处理某些性质时可能不够精确。
-
波函数方法(如 Hartree-Fock、MP2 和 CCSD(T))则通常提供更高的准确性,但计算成本更高,适用于需要高精度计算的情况。
-
混合方法(如 HSE 和 B3LYP)试图结合两者的优点,提供更好的性能。
-
G4MP2 和 QM7b 是应用于特定任务的高级方法和数据集,分别侧重于高精度计算和机器学习。
-
实验测量通常在某些情况下被认为比计算方法更可靠,尤其是在需要直接观察材料或分子行为时,但是某些实验技术的限制(如样品纯度、环境条件等)可能导致实验结果的偏差。
三、示例文件train.py
作者给出了一个示例文件供参考:train.py 是一个四保真模型的训练脚本 (PBE/GLLB-SC/HSE/SCAN)。在linux系统中可以直接运行脚本 runall.sh 来下载数据并且自动运行train.py
(拟合结果是一个最佳模型名为best_model.hdf5 ,其配置文件是 best_model.hdf5.json , 测试错误记录在 test_errors.txt 中,拟合日志记录将会被记录在 log.txt中)
train.py文件代码解释:
1.数据集:
ALL_FIDELITIES = ["pbe", "gllb-sc", "hse", "scan"]
TRAIN_FIDELITIES = ["pbe", "gllb-sc", "hse", "scan"]
VAL_FIDELITIES = ["gllb-sc", "hse", "scan"]
TEST_FIDELITIES = ["pbe", "gllb-sc", "hse", "scan"]
training data训练数据:PBE + GLLB-SC + HSE + SCAN数据集
validation data验证数据:GLLB-SC + HSE + SCAN (验证数据不包含PBE是因为,PBE数据集太大了,会导致模型偏向PBE的精准拟合)
test data测试数据:PBE + GLLB-SC + HSE + SCAN
训练集(Training Set):用于训练模型的数据。
验证集(Validation Set):用于调优和评估模型的数据,不参与训练。(用于评估模型、超参数调优、防止过拟合)
测试集(Test Set):用于最终评估模型性能的数据,确保模型的泛化能力。
2.一些参数设置
# 设置 epochs 的最大值为 1500
EPOCHS = 1500
# Random seed
SEED = 42
# Use the GPU with index 0. I use GTX 1080Ti, so one GPU is enough.By default, tensorflow will use all GPUs at maximum capability, this is not what we need.
GPU_INDEX = "0"
# 使用索引为0的GPU。作者使用GTX 1080TI,因此一个GPU就足够了。(默认情况下,TensorFlow将使用所有GPU)
-
机器学习中的一些概念
-
EPOCHS:整个训练数据集被用于训练模型的次数,在每一个 EPOCH 中,整个训练过程会被重复执行
-
随机初始化参数
-
前向传播:模型对某一个batch的训练样本进行预测,计算输出(eg:训练集有1000条数据,每个batch中有100条数据,那么这一轮的EPOCH中会包含10个batch。该计算步骤会对batch里的每一条数据进行计算)
-
损失计算:根据上一个步骤中的100个输出分别计算它们与真实值的损失,并使用某种损失函数。
eg:
-
反向传播:通过反向传播算法,模型调整其参数以减少损失。即计算损失相对于每个参数(即若干特征Wi和b)的梯度,然后更新参数
-
迭代:对训练数据集中剩余的batch进行重复这上述几个步骤,直至完成一个 EPOCH
-
-
-
为什么每次迭代学习会更改batch数据,而不是对某一个batch数据进行反复多次的迭代学习呢?
-
提高泛化能力,避免过度拟合:如果模型只对一个特定的 batch 数据进行多次更新,它可能会在这个 batch 上表现得很好,但对其他数据的泛化能力可能会下降。通过在每个迭代中使用不同的 batch,模型能够接触到更多样化的数据,从而学习到更一般化的特征
-
提高训练效率,快速收敛:通过多次使用不同的 batch,模型能够更快地接触到整个训练集,这能够加速收敛过程。尽早接触到各种样本可以让模型更快地调整其参数,以适应数据的整体分布
-
减小计算负担:在大规模深度学习模型训练中,通常希望在合理的时间内完成训练,因此使用不同的 batch 来更新参数更为高效。
-
seed:随机种子,为了控制随机性。随机种子值相同时,每次运行代码,将获得相同的随机数序列,确保实验结果一致。
-
可重复性:使用相同的Seed,可以复现他人的工作或自己之前的工作,使得模型训练和评估过程保持一致,使得结果能够被其他研究者验证和重现。
-
调试:在调试模型时,使用相同的 seed 可以更容易分析和理解模型的行为,因为可以确定所有的随机因素都是相同的。
-
比较实验:在进行不同模型或参数设置的比较时,使用相同的 seed 能保证所有实验在相同的数据划分和初始化条件下进行,有助于验证结果的有效性。
-
-
import os
# 是一行用于设置 NVIDIA CUDA 环境变量的代码,通常在使用深度学习框架(如 TensorFlow 或 PyTorch)时指定要使用的 GPU
os.environ["CUDA_VISIBLE_DEVICES"] = "0"
# CUDA:(Compute Unified Device Architecture)是 NVIDIA 提供的一个并行计算平台和编程模型,允许开发者利用 GPU 的强大性能进行通用计算。
# CUDA_VISIBLE_DEVICES 是一个环境变量,用于控制哪些 GPU 对 CUDA 程序可见。当运行深度学习程序时,默认情况下会使用所有可用的 GPU。但有时候,可能希望限制程序只使用某些特定的 GPU,以避免与其他用户或进程争夺资源
# 设置为 "0" 意味着程序只能访问第一个 GPU(如果存在多个 GPU)
# 如果想让程序使用多个 GPU,可以将它们的编号以逗号分隔,eg:os.environ["CUDA_VISIBLE_DEVICES"] = "0,1"
# 如果不想使用任何 GPU,可以将其设置为空字符串,这样CUDA 程序将只会使用 CPU 进行计算:os.environ["CUDA_VISIBLE_DEVICES"] = ""
3.建立模型
-
magnet介绍
-
MEGNet:是一种基于图神经网络的模型,专门设计用来处理材料科学中的晶体结构数据。它能够通过学习材料的图表示,捕捉材料的特性,并进行材料性质预测、稳定性分析等
-
晶体图表示:在MEGNet中,晶体结构被表示为图,其中原子作为节点,节点之间的连接(边)可以表示原子之间的相互作用或距离关系。CrystalGraph 类通常用于构建这种图表示
-
-
CrystalGraph 的用途
- 加载数据:从文件或其他源加载晶体结构数据。
- 创建图表示:将晶体结构转换为图的形式,以便输入到图神经网络中进行训练或推理。
- 提取特征:计算与晶体结构相关的各种特征,为后续模型训练做准备
## Graph converter
# GaussianDistance:是一个用于计算距离的转换器,将原子间的距离转换为高斯型相似性度量。它的输出用作图中边的特征
# centers:通过 np.linspace(0, 6, 100) 创建了100个均匀分布在0到6之间的点。这些点通常表示高斯分布的中心位置,代表不同的距离值。
# width:设置为 0.5 的宽度参数,控制高斯分布的宽度,影响到邻近原子间相互作用的强度和范围
# cutoff 参数设定了构建图时考虑的最大距离。在此例中,只有当两个原子之间的距离在cutoff范围内(小于或等于5.0)才会建立连接。即如果两个原子的距离超过 5.0,则它们不会被视为相互作用的节点。
crystal_graph = CrystalGraph(bond_converter=GaussianDistance(centers=np.linspace(0, 6, 100), width=0.5), cutoff=5.0)
# 使用该 crystal_graph 可以添加具体的晶体结构:crystal_graph.add_structure(structure) 添加某晶体结构
## model setup 创建了一个 MEGNet 模型实例,用于处理图形数据(如晶体结构)的预测或分类任务
model = MEGNetModel(
nfeat_edge=100, #这是边特征的维度。在图神经网络中,边可以表示不同原子之间的相互作用。这个参数定义了每条边将具有多少个特征
nfeat_global=None, #全局特征的维度。如果设置为 None,即不使用全局特征。全局特征一般用于表示整个图的特性,例如整个分子的能量或其他宏观性质。
ngvocal=len(TRAIN_FIDELITIES), #表示模型输出的类别有几种;len(TRAIN_FIDELITIES) 的结果是4,这意味着模型将需要处理四个不同的输出类别
global_embedding_dim=16,# 如果模型使用全局特征,那么这些全局特征会被嵌入到一个16维的向量空间中。这有助于将更复杂的数据压缩成较小的维度,从而使得它们在神经网络中的处理更加高效。
nblocks=3, # 网络中的块(block)数量。这通常表示图神经网络中的层数
nvocal=95,# 表示嵌入层(Embedding Layer)中输入特征的数量,表示模型的嵌入层将能够处理这95种不同的元素类型。换句话说,输入给嵌入层的特征数是95,每个特征代表一种元素。
npass=2, # 消息传递的次数;npass=2 表示在图的消息传递过程中进行了两次迭代,使得节点能够从其邻居处获取更深层次的信息,从而使得模型能够捕捉到更复杂的关系和模式。
graph_converter=crystal_graph,
lr=1e-3, # 学习率(learning rate),这是优化算法的重要超参数,控制着模型权重更新的步长
)
-
epoch和神经网络的层数 概念上的区别?
-
Epoch: 是与训练过程相关的一个参数,表示数据集被使用的次数,通常是在训练过程中调整的超参数。
-
层数: 是与模型结构相关的一个属性,表示网络的复杂性和深度,是设计网络时需要考虑的因素。
-
不需要为每一层单独定义epoch。epoch是针对整个训练过程的,是针对所有层的概念。
-
示例:假设有一个简单的三层神经网络,在训练过程中:
- 定义了每一层的结构。
- 决定将数据集用 10 epochs 进行训练。
- 在每个epoch中,所有层都会进行训练(接收输入、计算输出、计算损失,并通过反向传播更新其参数)。即每一层都训练过后才会进入下一个epoch,重复训练过程。
-
4.数据加载和处理
加载数据:
runall.sh:
#!/bin/bash
# download the zip file from figshare
echo "wget mp.2019.04.01.json.gz"
wget https://ndownloader.figshare.com/files/15108200
# move the downloaded file to get the correct filename
mv 15108200 mp.2019.04.01.json.gz
# unzip the file
echo "Unzip the file ... "
gunzip mp.2019.04.01.json.gz
# Model fitting
echo "Running training script"
nohup python train.py > log.txt&
## Structure data for all materials project materials 打开mp数据库中所有材料的结构数据
if not os.path.isfile("mp.2019.04.01.json"):
raise RuntimeError("Please download the data first! Use runall.sh in this directory if needed.")
with open("mp.2019.04.01.json") as f:
structure_data = {i["material_id"]: i["structure"] for i in json.load(f)}
print("All structures in mp.2019.04.01.json contain %d structures" % len(structure_data))
## Band gap data
with gzip.open("data_no_structs.json.gz", "rb") as f:
bandgap_data = json.loads(f.read()) # Python中的json.load()函数用于解析JSON文件并将其转换为相应的Python对象(例如字典或列表)。
# 下面这句代码中的*符号的作用:把列表推导式生成的list进行拆包(这个列表中包含若干个set集合),拆包后就去掉了list外壳只剩下若干个set集合了,接着用set.union()函数对这几个set集合进行合并生成一个大的set集合
useful_ids = set.union(*[set(bandgap_data[i].keys()) for i in ALL_FIDELITIES]) # mp ids that are used in training
print("Only %d structures are used" % len(useful_ids))
print("Calculating the graphs for all structures... this may take minutes.")
structure_data = {i: structure_data[i] for i in useful_ids}
structure_data = {i: crystal_graph.convert(Structure.from_str(j, fmt="cif")) for i, j in structure_data.items()}
# Structure.from_str(j, fmt="cif")是把一个CIF格式的晶体结构转换为一个Structure对象
# crystal_graph.convert()是把一个Structure对象转换为一个晶体图的形式
# i是mp_id ; j是mp_id对应的晶体结构图
## Generate graphs with fidelity information
graphs = []
targets = []
material_ids = []
for fidelity_id, fidelity in enumerate(ALL_FIDELITIES):
for mp_id in bandgap_data[fidelity]:
graph = deepcopy(structure_data[mp_id]) # deepcopy()深度复制改结构对象,防止修改操作对原始对象有影响
# The fidelity information is included here by changing the state attributes
# PBE: 0, GLLB-SC: 1, HSE: 2, SCAN: 3
graph["state"] = [fidelity_id]
graphs.append(graph)
targets.append(bandgap_data[fidelity][mp_id])
# the new id is of the form mp-id_fidelity, e.g., mp-1234_pbe
material_ids.append(f"{mp_id}_{fidelity}")
final_graphs = {i: j for i, j in zip(material_ids, graphs)}
final_targets = {i: j for i, j in zip(material_ids, targets)}
5.数据划分
# train:val:test = 8:1:1
fidelity_list = [i.split("_")[1] for i in material_ids]
train_val_ids, test_ids = train_test_split(
material_ids, stratify=fidelity_list, test_size=0.1, random_state=SEED)
# 在这一行中,整个数据集(material_ids)被划分为两个部分:train_val_ids(训练集和验证集的组合)和test_ids(测试集)。
# stratify参数确保不同类别在fidelity_list中的分布保持一致。
# test_size=0.1表示10%的数据将用于测试集
fidelity_list = [i.split("_")[1]
for i in train_val_ids] # 重新评估train_val_ids的保真度列表
train_ids, val_ids = train_test_split(
train_val_ids, stratify=fidelity_list, test_size=0.1 / 0.9, random_state=SEED)
# 此处test_size=0.1 / 0.9表示约11.1%的train_val_ids将分配给验证集
# remove pbe from validation
val_ids = [i for i in val_ids if not i.endswith("pbe")]
print("Train, val and test data sizes are ", len(train_ids), len(val_ids), len(test_ids))
# Get the train, val and test graph-target pairs
def get_graphs_targets(ids):
"""
Get graphs and targets list from the ids
Args:
ids (List): list of ids
Returns:
list of graphs and list of target values
"""
ids = [i for i in ids if i in final_graphs]
return [final_graphs[i] for i in ids], [final_targets[i] for i in ids]
train_graphs, train_targets = get_graphs_targets(train_ids)
val_graphs, val_targets = get_graphs_targets(val_ids)
6.模型训练
callbacks = [ReduceLRUponNan(patience=500), ManualStop()]
model.train_from_graphs(
train_graphs, train_targets, val_graphs, val_targets, epochs=EPOCHS, verbose=2, initial_epoch=0, callbacks=callbacks
)
这段代码实现了一个基于图形数据的模型训练过程,同时引入了学习率调整和手动停止的机制,以提高训练的稳定性与效率。通过设置这些回调,模型可以在面对潜在问题时更加灵活地响应,最终导致更好的训练成果。
回调函数定义
callbacks = [ReduceLRUponNan(patience=500), ManualStop()]
-
ReduceLRUponNan(patience=500): 这是一个回调函数,用于监控模型训练过程中的学习率调整。当训练过程中出现NaN(Not a Number)值时,它会减少学习率。patience=500表示如果在连续500个epoch内没有检测到有效的改进,就会触发学习率的降低。这可以帮助避免训练过程中的不稳定性,确保模型能够收敛。 -
ManualStop(): 这是另一个回调函数,通常用于手动停止训练。如果满足某些条件(如达到预设的性能指标),则可以提前结束训练。这有助于节省训练时间,防止过拟合或进行无效的训练。
模型训练
model.train_from_graphs(
train_graphs, train_targets, val_graphs, val_targets, epochs=EPOCHS, verbose=2, initial_epoch=0, callbacks=callbacks
)
model.train_from_graphs(...): 这是调用模型的方法来进行训练。名称表明它是从图形数据进行训练,适合处理图神经网络(Graph Neural Networks)等模型。
参数解释:
-
train_graphs: 训练集中的图形数据,通常是一组图的表示形式,它们将被输入到模型中进行训练。 -
train_targets: 对应于训练图形的目标标签,用于监督学习时的目标输出。 -
val_graphs: 验证集中的图形数据,用于在训练过程中评估模型的性能,以便监测是否发生了过拟合。 -
val_targets: 验证集对应的目标标签,用于与模型预测结果进行比较,从而计算验证损失和其他指标。 -
epochs=EPOCHS: 指定训练的总轮数(epoch)。 -
verbose=2: 设置日志显示的详细程度。verbose=2意味着在每个epoch结束后将输出更为详细的信息,包括训练和验证损失及其它指标,便于观察训练过程。 -
initial_epoch=0: 指定开始训练的初始epoch数。在恢复训练或继续之前的训练时,这个参数非常有用,但在新训练时通常设为0。(假设我们先前已经训练过模型,之后由于某种原因需要中断训练,例如资源不足、需修改模型结构或超参数等。在这种情况下,如果想要继续训练,我们就需要指定从哪个epoch开始继续训练。这时,initial_epoch 参数就不为0而是之前中断的那个epoch) -
callbacks=callbacks: 将之前定义的回调函数传递给模型训练方法。这使得在训练期间能够动态调整学习率和手动停止训练。
7.模型测试
-
模型训练的过程就是不断调整参数的过程。例如在一个线性回归模型中y=wx+b,w(权重)和b(偏置)就是模型的参数。训练数据被输入到模型中,模型根据当前的参数值产生预测结果,然后通过比较预测结果与真实结果之间的差异(损失函数)来对参数进行调整。这里的和就是待学习的参数,其中就是一种权重形式
-
模型权重是模型内部的一组参数。在神经网络中,这些参数通常以矩阵的形式存在
# load the best model with lowest validation error
files = glob("./callback/*.hdf5")
# glob 是 Python 的一个模块,用于查找符合特定模式的文件。在这里,"./callback/*.hdf5" 指的是在当前路径下的 callback 文件夹中查找以 .hdf5 为后缀名的文件。这通常用于存储训练好的模型权重
best_model = sorted(files, key=os.path.getctime)[-1]
# sorted 函数用于将 files 列表中的文件按创建时间进行排序。key=os.path.getctime 表示根据文件的创建时间来排序,从而确保最新创建的文件排在最后。
# [-1]: 通过索引 [-1] 获取排序后的列表中的最后一个元素,即最新创建的 .hdf5 文件。这个文件被假定为最佳模型权重
model.load_weights(best_model)
# 使用 load_weights 方法将之前找到的最新模型权重加载到 model 对象中。这使得模型可以恢复到使用该权重时的状态,通常用于继续训练或评估已经训练好的模型
model.save_model("best_model.hdf5") # 保存模型
# 计算模型对测试数据的预测值与真实目标值之间的平均绝对误差MAE
def evaluate(test_graphs, test_targets):
"""
Evaluate the test errors using test_graphs and test_targets
Args:
test_graphs (list): list of graphs
test_targets (list): list of target properties
Returns:
mean absolute errors
"""
# model.graph_converter 是一个对象,可能是负责将图形数据进行转换的工具。
# get_flat_data 方法可能会将输入的图和目标数据扁平化,以便后续处理。
test_data = model.graph_converter.get_flat_data(test_graphs, test_targets)
# *test_data:解包操作符: 星号 * 的使用表明 test_data 是一个可迭代对象,例如列表或元组,并且它的每个元素都会被解包为独立的参数。
# 创建一个 GraphBatchDistanceConvert 实例,其中包含扁平化后的图数据,并指定 用于距离计算的转换器 和 批量大小。这将使得后续处理能够高效地进行批量数据处理。
gen = GraphBatchDistanceConvert(*test_data, distance_converter=model.graph_converter.bond_converter, batch_size=128)
# 使用 model 对象中的 graph_converter 属性下的 bond_converter 来作为距离转换器。这表明在处理图形批次时,会采用与边特征相关的逻辑来计算节点间的距离或相似性。这是构建图神经网络模型时常见的一种做法,旨在更好地捕捉图中元素之间的关系
preds = []
trues = []
for i in range(len(gen)):
d = gen[i] # d代表当前批次,包含图数据和目标值的结构;d[0]: 当前批次的图数据。这可能包括图的特征、节点信息、边信息等;d[1]: 当前批次对应的真实目标值。
preds.extend(model.predict(d[0]).ravel().tolist())# 使用ravel() 将多维数组变为一维,然后使用tolist()转换为列表。
trues.extend(d[1].ravel().tolist()) # list.extend()是列表对象的一个方法,用于将一个可迭代对象中的所有元素添加到列表的末尾
return np.mean(np.abs(np.array(preds) - np.array(trues)))
# Calculate the errors on each fidelity
test_errors = []
for fidelity in TEST_FIDELITIES:
test_ids_fidelity = [i for i in test_ids if i.endswith(fidelity)]
test_graphs, test_targets = get_graphs_targets(test_ids_fidelity)
test_error = evaluate(test_graphs, test_targets)
test_errors.append(test_error)
# Save errors
with open("test_errors.txt", "w") as f:
line = [f"{i}: {j:.3f} eV\n" for i, j in zip(TEST_FIDELITIES, test_errors)]
# 使用了列表推导式来生成一个包含字符串的列表 line。这个列表中的每个元素都是一个格式化后的字符串
# zip(TEST_FIDELITIES, test_errors) 会将 TEST_FIDELITIES 列表和 test_errors 列表中的元素一一配对,生成一个元组的迭代器
f.write("".join(line)) # "".join(line) 将列表 line 中的所有字符串合并成一个单一的字符串,以便于一次性写入文件
四、运行train.py
因为下载数据集mp.2019.04.01.json.gz的网址不可达,因此手动下载数据集并手动解压在train.py同一目录下,接着手动运行train.py
nohup python train.py > output.log 2>&1 &
-
nohup 会将输出(标准输出和错误输出)重定向到一个名为 nohup.out 的文件。如果您想指定输出文件,可以使用重定向操作符,
> output.log将标准输出重定向到 output.log 文件 -
2>&1:将标准错误输出(文件描述符 2)也重定向到标准输出(文件描述符 1),从而使所有输出都在同一个文件中
(1)第一次运行结果:
从output.log日志来看,训练过程中出现了两个主要问题:
INFO:megnet.callbacks:
Epoch 00016: val_mae improved from 0.68293 to 0.67450, saving model to callback/val_mae_00016_0.674496.hdf5
Epoch 16/1500
383/383 - 311s - loss: 0.6855
INFO:megnet.callbacks:Nan loss found!
Epoch 17/1500
Traceback (most recent call last):
......
File "/home/ubuntu/anaconda3/envs/myenv2/lib/python3.7/site-packages/tensorflow_core/python/keras/engine/training.py", line 181, in load_weights
return super(Model, self).load_weights(filepath, by_name)
File "/home/ubuntu/anaconda3/envs/myenv2/lib/python3.7/site-packages/tensorflow_core/python/keras/engine/network.py", line 1177, in load_weights
saving.load_weights_from_hdf5_group(f, self.layers)
File "/home/ubuntu/anaconda3/envs/myenv2/lib/python3.7/site-packages/tensorflow_core/python/keras/saving/hdf5_format.py", line 651, in load_weights_from_hdf5_group
original_keras_version = f.attrs['keras_version'].decode('utf8')
AttributeError: 'str' object has no attribute 'decode'
-
(1) NaN Loss:这表明模型在第16epoch 的训练结束后出现了NaN(不是一个数字)的损失,这意味着在这一轮训练中,损失值变成了NaN(不是一个数字)。可能的原因包括:
- 学习率过高,导致梯度爆炸(在训练深度学习模型时,梯度的值变得非常大,从而导致权重更新时产生极大的变化。这通常会导致模型不稳定,损失函数发散,从而导致训练失败),大梯度更新可能导致权重变得不稳定,从而产生 NaN
- 输入数据中存在无效值(如 NaN 或无限大)。
- 模型的不稳定性,可能是架构设置不当或参数初始化问题
-
(2)AttributeError: 随后,程序因尝试加载权重时遇到了AttributeError而终止。这个错误是由于代码试图访问HDF5文件中的属性,但由于某种原因,该属性的类型不符合预期(字符串而不是字节对象)。可能的原因包括:
-
Keras版本不匹配:如果你的模型是在不同版本的Keras/TensorFlow中训练并保存的,而现在使用的是另一个版本,可能会导致加载权重时出现不兼容的问题。确保使用相同版本的库。
-
HDF5文件的结构问题:如果HDF5文件没有正确保存(例如,由于未能在训练中正确结束),这可能导致无法加载权重。确保在每次保存模型时都没有错误。
-
修改Keras代码:如果你在Keras的实现中做了自定义改变,确保这些改变没有影响到模型的权重保存和加载过程。
-
因此首先尝试修改学习率来解决,把原来的lr=1e-3改成lr=1e-4(从0.001调整成0.0001)
model = MEGNetModel(
nfeat_edge=100,
nfeat_global=None,
ngvocal=len(TRAIN_FIDELITIES),
global_embedding_dim=16,
nblocks=3,
nvocal=95,
npass=2,
graph_converter=crystal_graph,
lr=1e-4,
)
(2)第二次运行结果:
重新执行nohup python train.py > output2.log 2>&1 &

在尝试修改学习率为0.00001
(3)第三次运行结果:
重新执行nohup python train.py > output3.log 2>&1 &
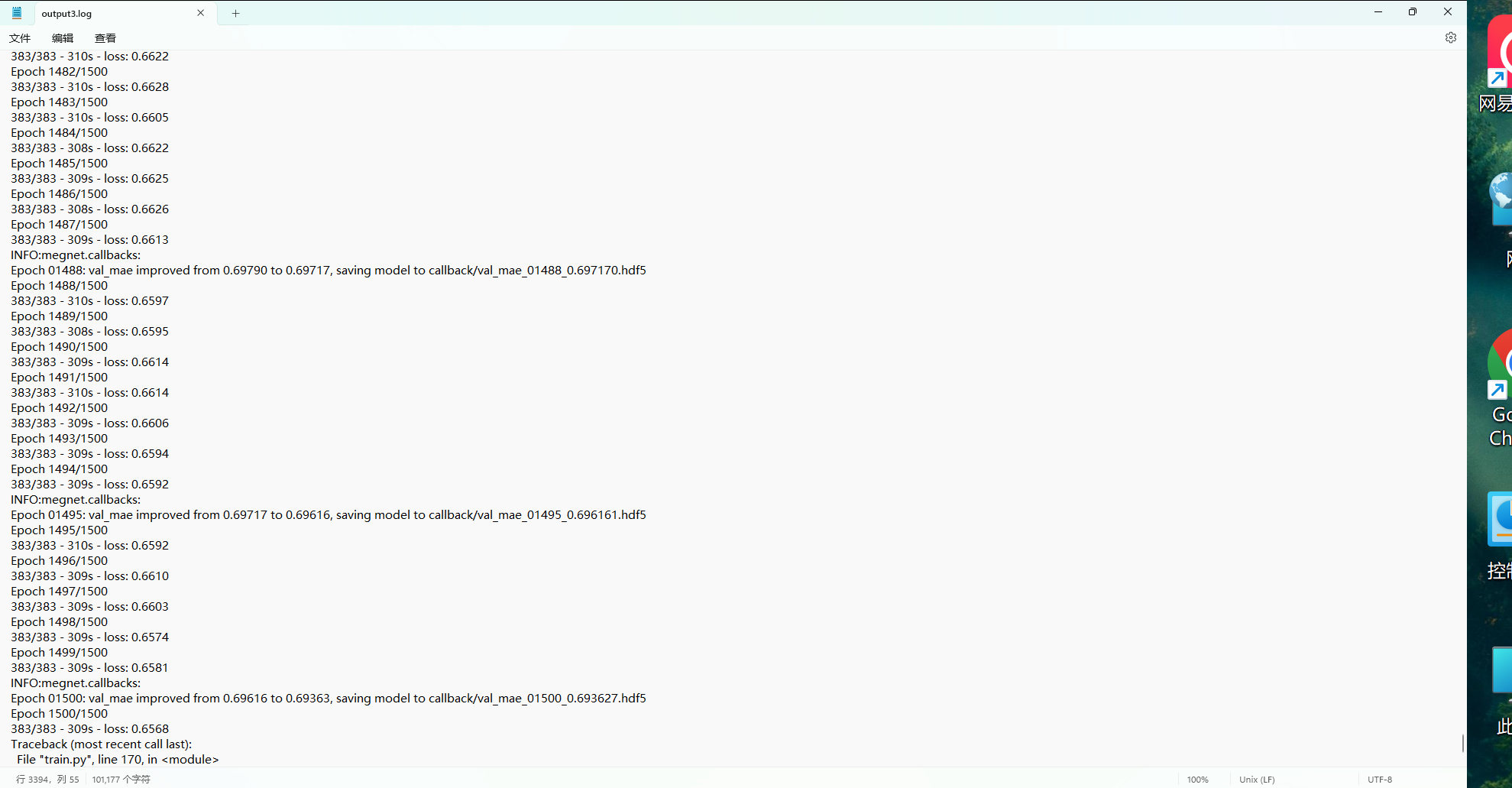
成功完成了1500个epochs训练过程,得到了最佳模型val_mae_01500_0.693627.hdf5
(4)问题:
train.py中的model testing代码并没有执行,也没有生成test_errors.txt文件。
可能是因为模型加载失败而中断执行,但是加载权重代码中没有写try except块,因此在输出文件中没有异常提示。
因此修改model testing部分的代码如下:
#原来
model.load_weights(best_model)
model.save_model("best_model.hdf5")
#修改的部分
try:
model.load_weights(best_model)
except Exception as e:
print(f"加载权重时出错: {e}") #如果加载权重失败,可以添加异常处理来捕捉并处理错误
model.save("best_model.hdf5") # 使用 model.save() 方法来保存模型,而不是 save_model()
-
因为数据集的划分是随机的,所以上一次训练出来的模型值可用上一次划分出的测试集进行评估才准确,然而上一次test过程出错没有运行成功,并且测试集的数据也没有单独保存,因此如果想要查看测试即的情况只能重新划分数据重新训练再重新测试模型。
-
但是,因为作者再数据划分时设置了固定的SEED值,因此重新划分后,数据集的划分情况将会一模一样,代码如下:
# Data splits
# train:val:test = 8:1:1
# 第一个划分:将 material_ids 划分为训练+验证集(train_val_ids)和测试集(test_ids),其中测试集占总数据的 10%
fidelity_list = [i.split("_")[1] for i in material_ids]
train_val_ids, test_ids = train_test_split(material_ids, stratify=fidelity_list, test_size=0.1, random_state=SEED)
# 第二个划分:再从训练+验证集中进一步划分为训练集(train_ids)和验证集(val_ids),验证集占训练+验证集的 10%。
fidelity_list = [i.split("_")[1] for i in train_val_ids]
train_ids, val_ids = train_test_split(train_val_ids, stratify=fidelity_list, test_size=0.1 / 0.9, random_state=SEED)
# remove pbe from validation
val_ids = [i for i in val_ids if not i.endswith("pbe")]
print("Train, val and test data sizes are ", len(train_ids), len(val_ids), len(test_ids))
因此,解决方案就是运行model_testing代码(复制train.py文件,剔除模型训练的代码,修改部分模型测试的代码)
model_testing:
from sklearn.model_selection import train_test_split
from megnet.models import MEGNetModel
from megnet.data.graph import GaussianDistance, GraphBatchDistanceConvert
from megnet.data.crystal import CrystalGraph
from megnet.callbacks import ManualStop, ReduceLRUponNan
from pymatgen.core import Structure
import numpy as np
from glob import glob
from copy import deepcopy
import os
import json
import gzip
import tensorflow as tf
ALL_FIDELITIES = ["pbe", "gllb-sc", "hse", "scan"]
TRAIN_FIDELITIES = ["pbe", "gllb-sc", "hse", "scan"]
VAL_FIDELITIES = ["gllb-sc", "hse", "scan"]
TEST_FIDELITIES = ["pbe", "gllb-sc", "hse", "scan"]
# Random seed
SEED = 42
# 2. Model construction
# Graph converter
crystal_graph = CrystalGraph(bond_converter=GaussianDistance(
centers=np.linspace(0, 6, 100), width=0.5), cutoff=5.0)
# model setup
model = MEGNetModel(
nfeat_edge=100,
nfeat_global=None,
ngvocal=len(TRAIN_FIDELITIES),
global_embedding_dim=16,
nblocks=3,
nvocal=95,
npass=2,
graph_converter=crystal_graph,
lr=1e-5,
)
# 3. Data loading and processing
# load data
# Structure data for all materials project materials
if not os.path.isfile("mp.2019.04.01.json"):
raise RuntimeError(
"Please download the data first! Use runall.sh in this directory if needed.")
with open("mp.2019.04.01.json") as f:
structure_data = {i["material_id"]: i["structure"] for i in json.load(f)}
print("All structures in mp.2019.04.01.json contain %d structures" %
len(structure_data))
# Band gap data
with gzip.open("data_no_structs.json.gz", "rb") as f:
bandgap_data = json.loads(f.read())
# mp ids that are used in training
useful_ids = set.union(*[set(bandgap_data[i].keys()) for i in ALL_FIDELITIES])
print("Only %d structures are used" % len(useful_ids))
print("Calculating the graphs for all structures... this may take minutes.")
structure_data = {i: structure_data[i] for i in useful_ids}
structure_data = {i: crystal_graph.convert(Structure.from_str(
j, fmt="cif")) for i, j in structure_data.items()}
# Generate graphs with fidelity information
graphs = []
targets = []
material_ids = []
for fidelity_id, fidelity in enumerate(ALL_FIDELITIES):
for mp_id in bandgap_data[fidelity]:
graph = deepcopy(structure_data[mp_id])
# The fidelity information is included here by changing the state attributes
# PBE: 0, GLLB-SC: 1, HSE: 2, SCAN: 3
graph["state"] = [fidelity_id]
graphs.append(graph)
targets.append(bandgap_data[fidelity][mp_id])
# the new id is of the form mp-id_fidelity, e.g., mp-1234_pbe
material_ids.append(f"{mp_id}_{fidelity}")
final_graphs = {i: j for i, j in zip(material_ids, graphs)}
final_targets = {i: j for i, j in zip(material_ids, targets)}
# 4. Data splits
# train:val:test = 8:1:1
fidelity_list = [i.split("_")[1] for i in material_ids]
train_val_ids, test_ids = train_test_split(material_ids, stratify=fidelity_list, test_size=0.1, random_state=SEED)
fidelity_list = [i.split("_")[1] for i in train_val_ids]
train_ids, val_ids = train_test_split(train_val_ids, stratify=fidelity_list, test_size=0.1 / 0.9, random_state=SEED)
# remove pbe from validation
val_ids = [i for i in val_ids if not i.endswith("pbe")]
print("Train, val and test data sizes are ", len(
train_ids), len(val_ids), len(test_ids))
# Get the train, val and test graph-target pairs
def get_graphs_targets(ids):
"""
Get graphs and targets list from the ids
Args:
ids (List): list of ids
Returns:
list of graphs and list of target values
"""
ids = [i for i in ids if i in final_graphs]
return [final_graphs[i] for i in ids], [final_targets[i] for i in ids]
train_graphs, train_targets = get_graphs_targets(train_ids)
val_graphs, val_targets = get_graphs_targets(val_ids)
# 6. Model testing
# load the best model with lowest validation error
files = glob("./callback/*.hdf5")
best_model = sorted(files, key=os.path.getctime)[-1]
try:
model.load_weights(best_model)
except Exception as e:
print(f"加载权重时出错: {e}") #如果加载权重失败,可以添加异常处理来捕捉并处理错误
model.save("best_model.hdf5") # 使用 model.save() 方法来保存模型,而不是 save_model()
def evaluate(test_graphs, test_targets):
"""
Evaluate the test errors using test_graphs and test_targets
Args:
test_graphs (list): list of graphs
test_targets (list): list of target properties
Returns:
mean absolute errors
"""
test_data = model.graph_converter.get_flat_data(test_graphs, test_targets)
gen = GraphBatchDistanceConvert(
*test_data, distance_converter=model.graph_converter.bond_converter, batch_size=128)
preds = []
trues = []
for i in range(len(gen)):
d = gen[i]
preds.extend(model.predict(d[0]).ravel().tolist())
trues.extend(d[1].ravel().tolist())
return np.mean(np.abs(np.array(preds) - np.array(trues)))
# Calculate the errors on each fidelity
test_errors = []
for fidelity in TEST_FIDELITIES:
test_ids_fidelity = [i for i in test_ids if i.endswith(fidelity)]
test_graphs, test_targets = get_graphs_targets(test_ids_fidelity)
test_error = evaluate(test_graphs, test_targets)
test_errors.append(test_error)
# Save errors
with open("test_errors.txt", "w") as f:
line = [f"{i}: {j:.3f} eV\n" for i, j in zip(TEST_FIDELITIES, test_errors)]
f.write("".join(line))
运行:

结果:
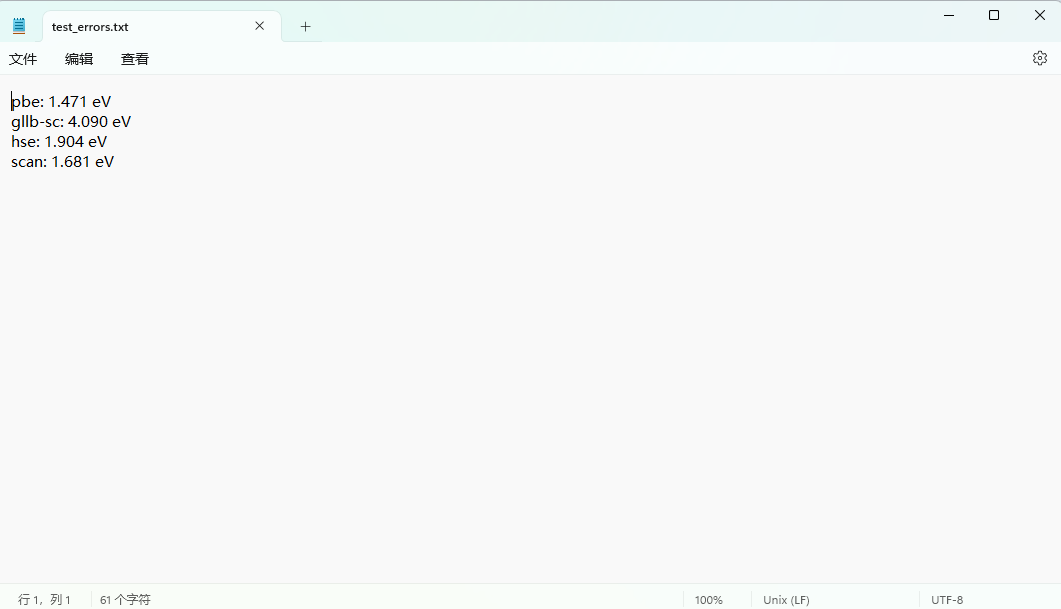
五、论文内容
1. 论文概述
主要介绍了多保真度图网络(multi - fidelity graph networks)在材料科学中的应用,通过结合不同保真度的数据来准确预测材料的性质,解决了高保真数据稀缺的问题,并展示了其在有序和无序材料中的通用性。
2. 研究背景
-
材料性质预测的挑战:
- 从原子排列预测材料性质是材料科学的基本目标。虽然机器学习为材料性质预测提供了新方法,但受高保真数据稀缺的限制。
- 从头算计算虽可靠,但计算成本高、可扩展性差,且更准确的方法成本更高。监督机器学习对其进行建模虽受关注,但是难以获取足够多的训练数据。
-
多保真度模型的潜在优势:
- 多保真度模型结合低保真和高保真数据,可解决数据不足的问题。以往相关工作存在局限,如训练复杂度高、限于特定性质和结构、难以扩展到多种保真度或要求数据同质。
3. 多保真度图网络方法
-
图网络表示:
- 材料以图网络表示,原子为节点,键为边,还包含结构无关的全局状态。输入的原子特征是嵌入的原子序数,键特征是高斯扩展距离,数据保真度编码为整数作为状态属性。
通过一系列图卷积层构建模型,依次更新原子、键和状态特征,最后读出潜在特征并传入神经网络进行性质预测。
- 材料以图网络表示,原子为节点,键为边,还包含结构无关的全局状态。输入的原子特征是嵌入的原子序数,键特征是高斯扩展距离,数据保真度编码为整数作为状态属性。
-
数据收集与处理:
- 收集了不同保真度的晶体带隙数据,包括 PBE、GLLB-SC、SCAN、HSE方法的计算数据,以及实验测量的数据。对数据进行过滤和处理,将其随机划分为训练、验证和测试集。
4. 实验结果
-
有序结构的带隙预测:
- 首先开发了单保真度图网络模型,发现其MAE与数据集大小和平均绝对偏差有关。多保真度模型利用 PBE 数据集和其他保真度数据,显著降低了 MAE,提高了预测准确性。
- 研究不同保真度组合的模型,发现包含PBE数据的多保真度模型效果更好,且增加保真度数量可提高模型一致性。多保真度图网络模型优于以往的机器学习模型和其他替代方法。
- 通过可视化潜在结构特征,发现包含大量低保真PBE数据有助于学习更好的潜在结构特征,从而提高高保真预测的准确性。
-
无序材料的带隙预测:
- 多保真度图网络架构为无序晶体的性质预测提供了框架。利用学习到的元素嵌入来表示无序位点,在无序晶体带隙预测上取得了较好的效果,且通过重新训练可进一步提高准确性。
- 通过对一些无序材料体系的带隙工程数据进行验证,表明模型能很好地再现定性趋势,并在多数体系中达到近定量的准确性。
5. 结论
-
方法的有效性和通用性:
- 多保真度图网络能够利用大量低成本的低保真计算数据有效地学习潜在结构特征,从而大幅提高对更昂贵的计算方法和实验的预测准确性。该框架具有通用性,不仅适用于晶体带隙预测,也适用于分子和其他材料性质的预测。
-
对无序材料的意义:
- 能够预测无序材料的带隙,表明图网络模型中学习到的元素嵌入以一种自然的方式编码了化学信息,为处理无序材料提供了一种替代方法,解决了传统机器学习模型和从头算计算在处理无序材料时的困难。
-
局限性和未来方向:
- 方法依赖于大量的低保真数据集来学习有效的潜在结构表示,对于一些性质可能无法获得足够大量的低保真数据。在这种情况下,转移学习方法可能更合适。未来研究可进一步探索如何在低保真数据有限的情况下提高模型性能。
6.涉及到的迁移学习
-
文中通过将大量的低保真度PBE数据训练得到的模型的图卷积层固定(即:对于已经训练好的PBE模型中的图卷积层的参数不再进行更新。这些参数保持其在PBE模型训练结束时的值),然后使用更高保真度的数据重新训练最终层来实现迁移学习。希望能够在这个基础上,使用更高保真度的数据来进一步优化模型对材料性质的预测能力,主要是通过重新训练模型的最终层来适应更高保真度数据的特点和要求。
-
迁移学习本质上是一种双保真度方法,需要两步训练过程。同时,它在处理某些数据集(如实验数据集)时效果不如一些多保真度图网络模型。例如,它在实验数据集上的误差高于多保真度模型,这表明多保真度图网络架构能够更好地捕捉不同保真度数据集之间的复杂关系。eg:在实验数据集上,迁移学习模型的 MAE 为 0.47 eV,而 2 - fi PBE/Exp 模型的 MAE 为 0.40 eV;以及表现最好的 PBE/GLLB - SC/HSE/Exp 的 4 - fi 模型比迁移学习模型性能更好。
6.megnet起到了什么作用呢?
-
具体来说,MEGNet 为多保真度图网络提供了基础的图神经网络架构和方法,而多保真度图网络则在此基础上增加了对多保真度数据的处理和利用能力。
-
megnet只是一个模型框架,而不是一个已经训练好的模型。本文并没有利用迁移学习来构造多保真模型,而是通过创新的多保真度图网络模型的训练方式(利用多保真数据集从头开始训练模型),而非依赖现有的模型迁移学习。

 浙公网安备 33010602011771号
浙公网安备 33010602011771号