中科院魏同镇教授解读韩春雨副教授基因编辑技术‘’NgAgo-gDNA‘’
韩春雨最新Nature Biotechnology文章详细解读
转自: 科学网—韩春雨最新Nature Biotechnology文章详细解读 - 魏同镇的博文
- 5月2日,Nature系列顶尖刊物_《Nature Biotechnology》在线发表了河北科技大学生物科学与工程学院青年教师韩春雨副教授题为“DNA-guided genome editing using the Natronobacterium gregoryi Argonaute”_的研究成果。
- 《Nature Biotechnology》主要发表生物技术领域的原始创新性研究成果,2015年JCR影响因子为41.514,已超过主刊《Nature》(41.456)。
![http://blog.sciencenet.cn/data/attachment/album/201605/11/093203dy4vv14h4ffsufmh.png]()


-
简单地说,作者发明了一种新的基因编辑技术(NgAgo-gDNA),适合在人类细胞中基因组编辑,不同于已有最时兴的技术(CRISPR-Cas9)。后者通过RNA寻找替换序列,而新技术通过DNA作为介导寻找替换目标。
-
NgAgo是Natronobacterium gregoryi Argonaute这一短语的简称。韩春雨团队就是利用格氏嗜盐碱杆菌(Natronobacterium gregoryi)的Argonaute实现了DNA引导的基因组编辑,并发现NgAgo作为一种DNA介导的核酸内切酶,适合在人体细胞中进行基因组编辑。
-
Argonaute作为一个核酸内切酶家族,最早由荷兰瓦赫宁恩大学(Wageningen University) 的约翰-范德欧斯特(John van der Oost)研究组证明这一家族的同源蛋白酶活,可以有效地利用5’磷酸化的ssDNA作为短介质,去相对精准地切割基因组靶点。然而无论是来源于Thermus thermophilus的Argonaute(TtAgo)还是来源于Pyrococcus furiosus的Argonaute(PfAgo)都需要在>65℃的条件下反应,这就限制了其在哺乳动物中应用的可能性。于是作者利用NCBI在线工具PSI-BLAST比对与TtAgo和PfAgo同源性高的可能蛋白,最终作者锁定了来自于N.gregoryi SP2的Argonaute——NgAgo。
-
首先,作者想确认NgAgo是否能利用ssDNA(single-stranded DNA)作为向导。
-
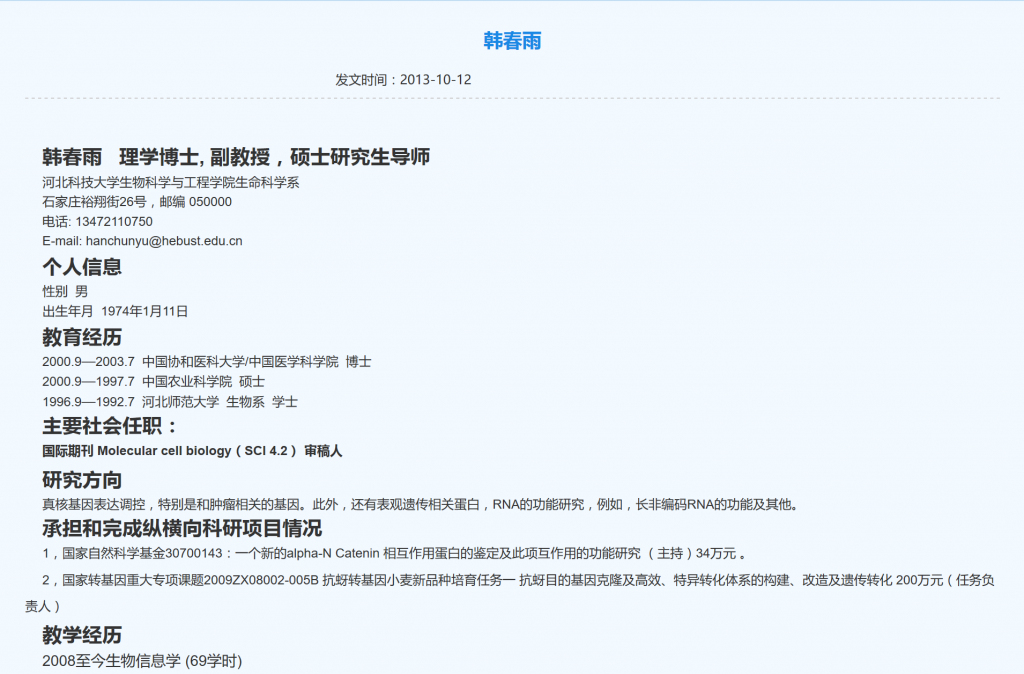
_E.coli_内既有5’磷酸化的ssRNAs也有相应的ssDNAs,所以作者从E.coli纯化出NgAgo,然后使用相应的Nuclease来消化。我们从Fig.1a可以看出,加入RNase A对NgAgo结合的Nucleotide无影响,而加入DNase I后,Nucleotide都被降解了,这说明NgAgo结合的是DNA而不是RNA。
-
下一步自然是要证明NgAgo可以在37℃下降解Target了,如果不可以就只能跟TtAgo和PfAgo一样的命运了。不过作者运气不错,NgAgo确实可以在37℃条件下利用5’磷酸化的ssDNA来剪切DNA,看看作者是如何设计实验的吧。
-
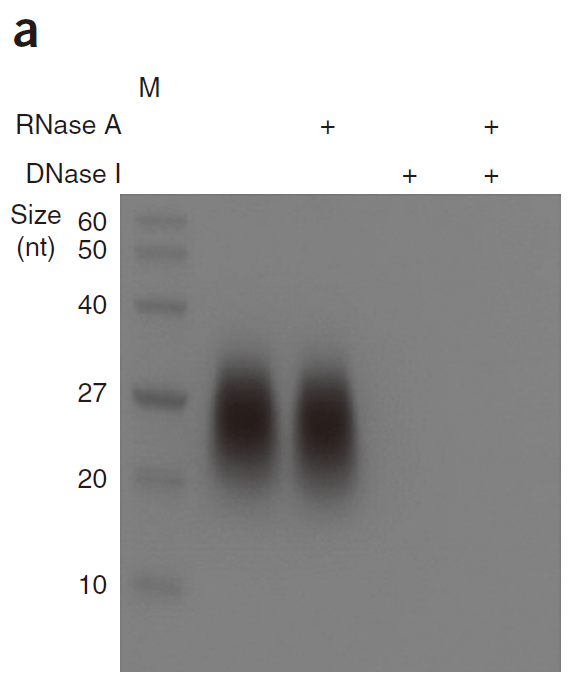
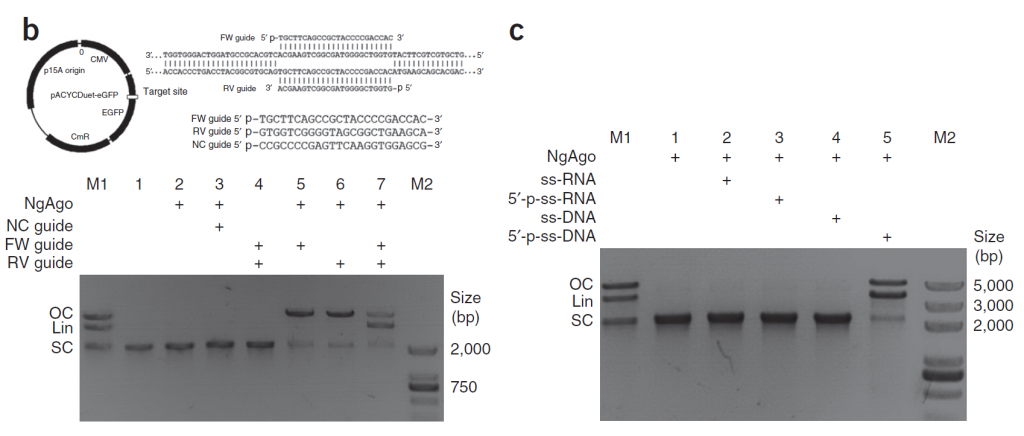
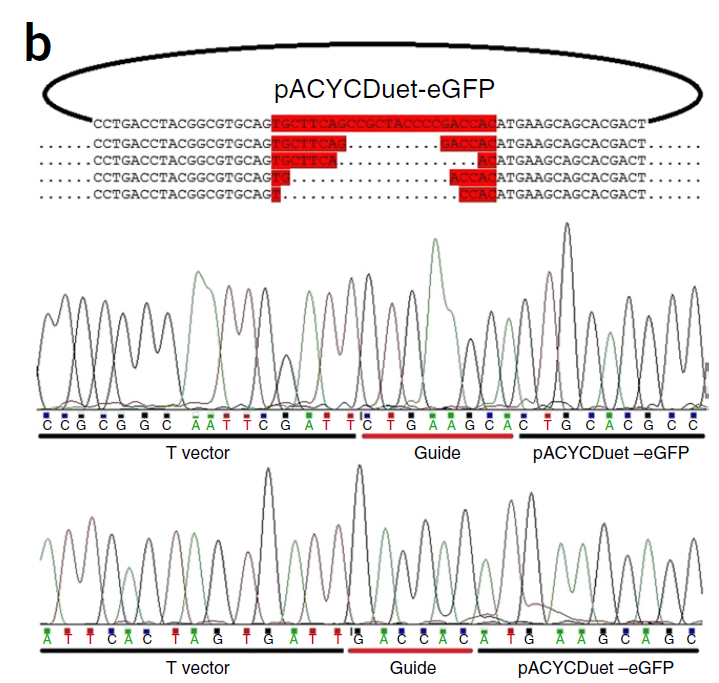
作者设计了三条24个Nucleotide长度的5’磷酸化的ssDNA guides,其中两个为互补并靶向质粒pACYCDuet-eGFP(‘FW’和‘RV’),还有一个是随机序列并无靶标的对照(noncomplementary guide‘NC’)。我们从Fig.1b上可以看出,只有FW和RV与NgAgo共转时才可以剪切相应质粒。作者还设计了相应的有无5’磷酸化的RNA和DNA,发现只有5'磷酸化的DNA才可以介导NgAgo的剪切。(Fig.1c)
- 好了,本篇文章大的基础成立了,这可是这篇文章的关键所在,大的前提如果不成立,一切就然并卵了。
- 接下来自然是探索NgAgo能不能在哺乳动物里进行基因组编辑了。
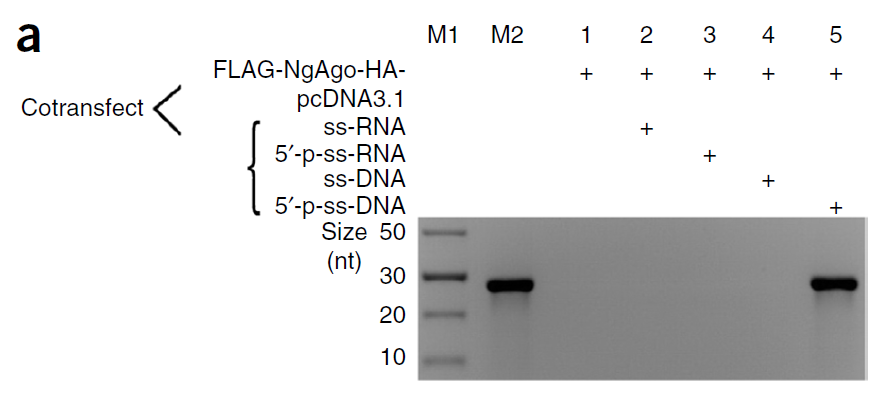
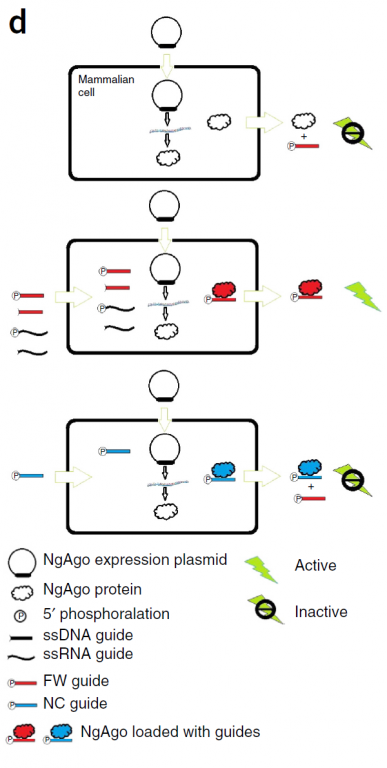
- 结果发现NgAgo在293T里并没有结合任何核酸分子(Fig.2a)。这说明至少在293T里是没有5'-p-ssDNA与NgAgo结合,也就减少了脱靶的可能。而且在293T里也只与5'-p-ssDNA结合。
- 之后发现先转入NgAgo,24h后再转入5'-p-ssDNA,NgAgo-ssDNA结合明显减少(Fig.2b)。

- 这说明NgAgo表达后短时间内就发生了NgAgo-ssDNA的结合。
- 从293T纯化出的NgAgo只有在55℃的时候才可以与5'-p-ssDNA结合(Fig.2c)。
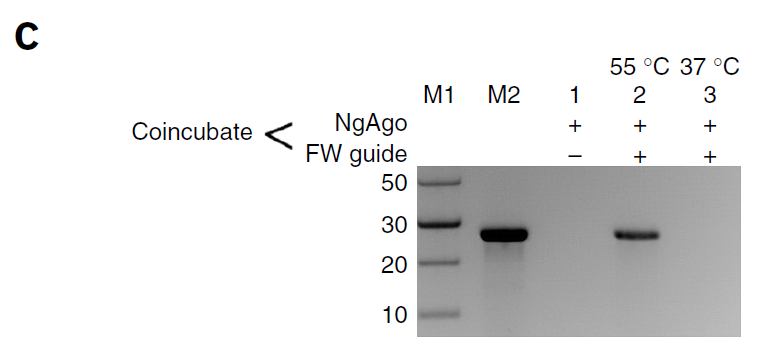
- 进一步的实验表明,只有从共转了NgAgo和相应5'-p-ssDNA的才可以对靶标进行剪切,而将从293T里纯化出的NgAgo与5'-p-ssDNA在体外孵育则无法发挥作用。并且从共转NC组的293T里纯化出的NgAgo再与FW孵育也无法剪切靶标(Fig.S1)。
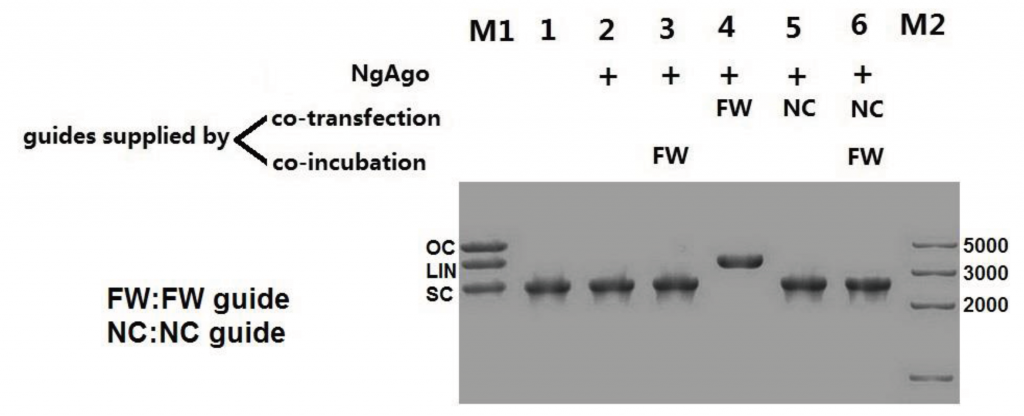
- 以上实验说明了,NgAgo一旦结合了5'-p-ssDNA就不会再与其他ssDNA结合。这也减少了其脱靶效应。
- 测序发现,NgAgo与TtAgo一样,都是在靶标位置移走部分核酸。为了排除NgAgo是一种核酸外切酶,作者将孵育时间延长到72h,但并未检测到靶标的降解(Fig.3a)。
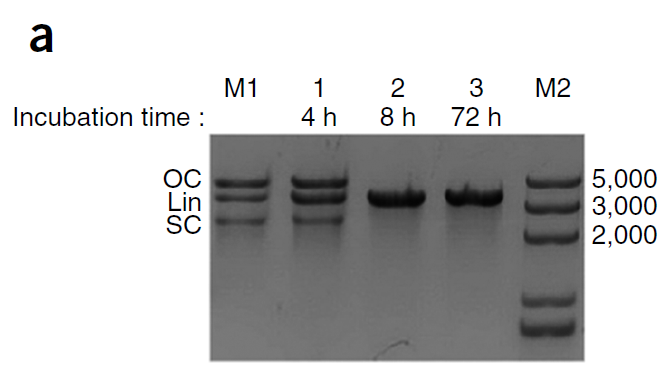
- 对孵育了72h之后的产物进行测序,发现在靶标位置内有1-20nt被随机移除了(Fig.3b)。
-
以上实验说明NgAgo并不是一种核酸外切酶。
-
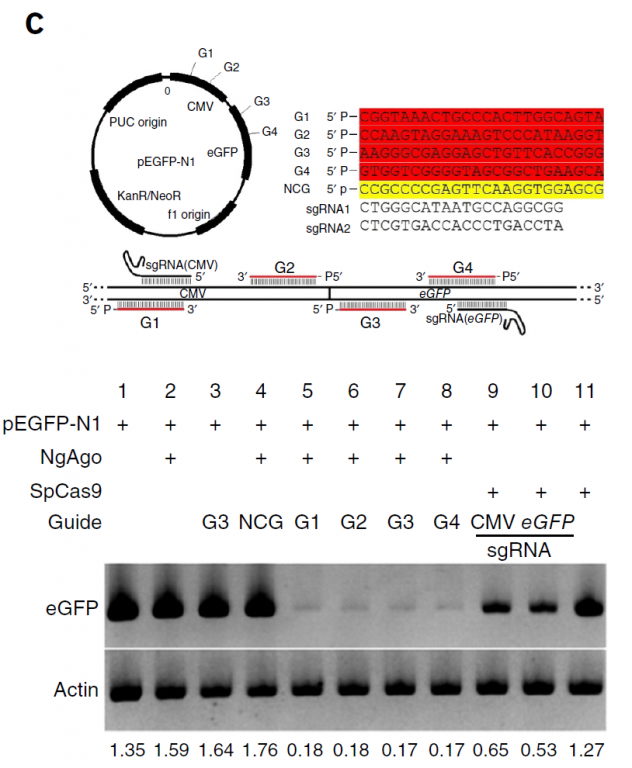
作者又设计了一个检测效率的系统。大体是设计了靶向pEGFP-N1质粒中的CMV或者eGFP的ssDNA guides,从而可以通过检测eGFP来对比其与Cas9的效率高低,结果发现NgAgo并不比Cas9差(Fig.3c)。从该图上看貌似比Cas9效率还要高,但是没有进一步的数据,我们也无法得出这样的结论,作者的结论也是很审慎,只是说该系统也如Cas9那样有效。

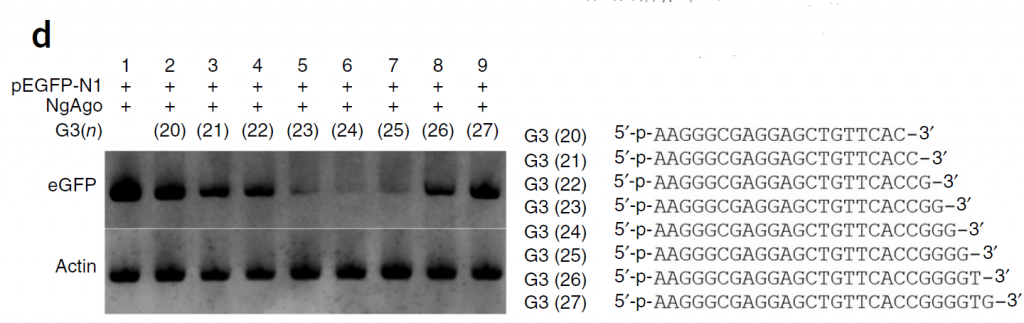
- 作者又利用上述系统将NgAgo需要的gDNA锁定在24nt(Fig.3d)
-
接下来,作者对NgAgo系统是否能编辑内源人类基因组进行了探索。
-
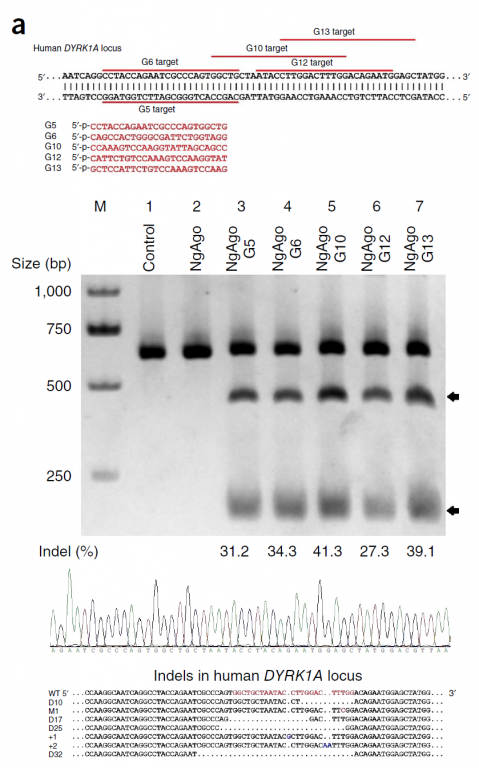
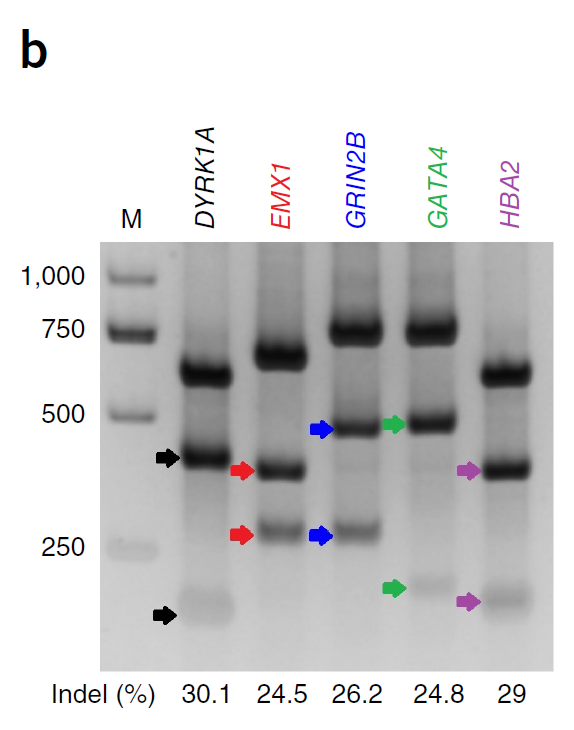
作者设计了针对人DYRK1A基因的gDNAs,结果发现该系统也是非常有效的(Fig.4a)。
- 之后又检测了针对八个不同基因的47条guides,发现效果均不错(Fig.4b)。
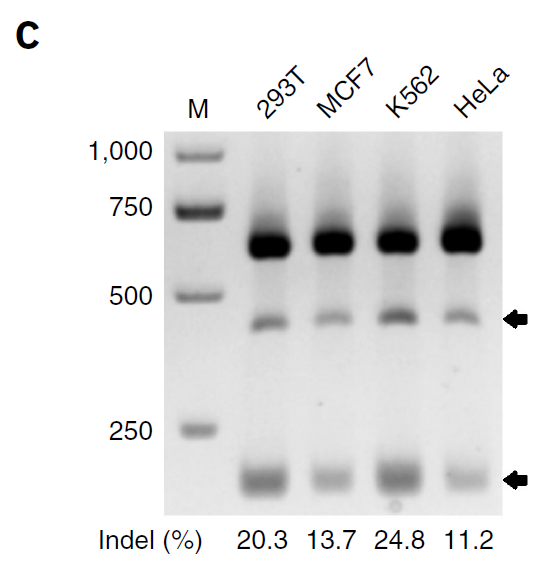
- 在293T、MCF7、K562和Hela细胞里也取得不错的结果(Fig.4c)
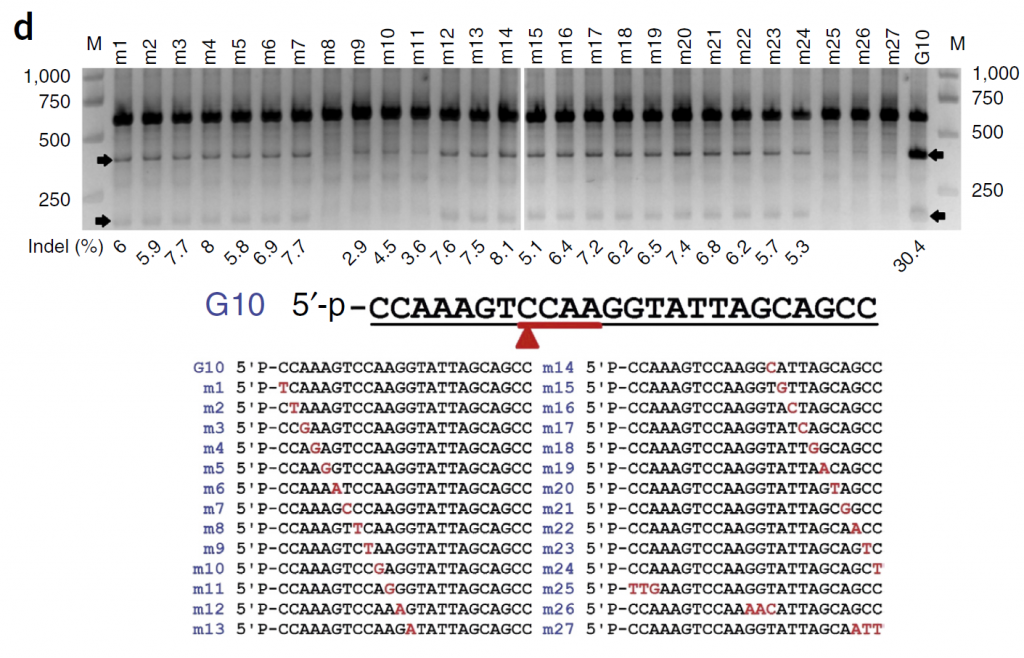
- 作者之后又测试了错配对NgAgo效率的影响,发现错配一个碱基平均可以减少73-100%的效率,P8-11位最为关键。而当有三个碱基错配的时候,NgAgo的作用基本消失(Fig.4d)。而Cas9系统最高可以容忍5个碱基的错配,所以这也是该系统的牛逼之处。
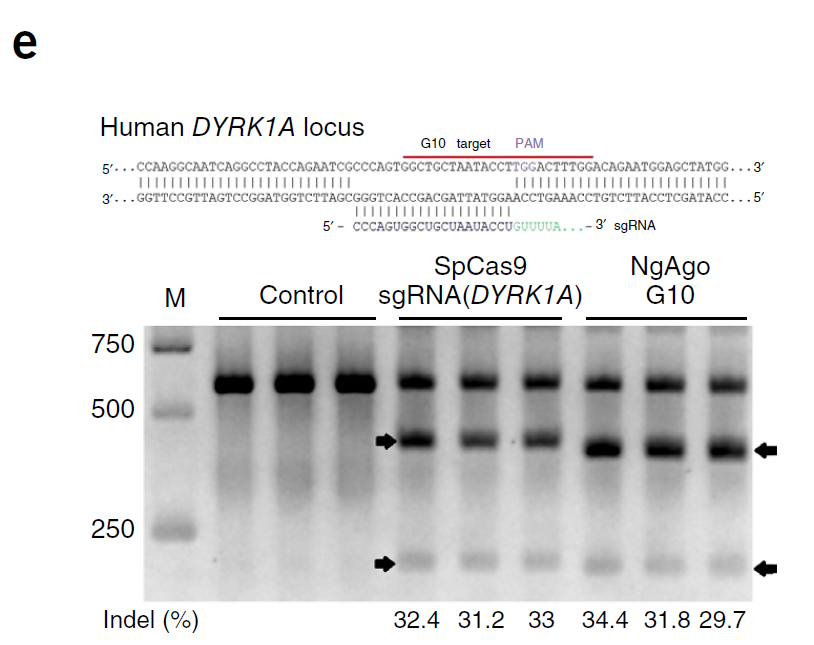
- 跟Cas9系统对比发现,效率有的一拼哦~(Fig.4e)
-
NgAgo系统对(G+C)富集区效率同样喜人(Fig.4f)。
![http://blog.sciencenet.cn/data/attachment/album/201605/11/093218wllqjonegnk1qlj1.png]()
-
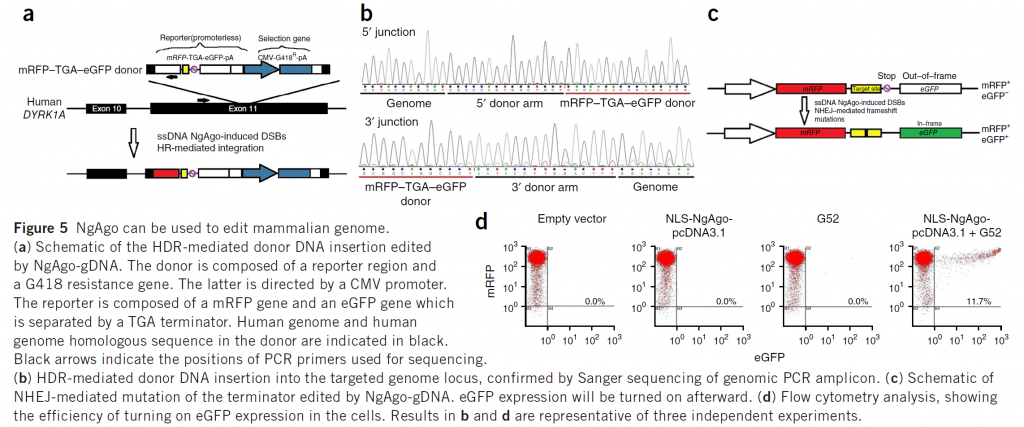
Cas9可不止删除功能,要想把其拉下神坛,NgAgo功能得可以与Cas9比肩才行,所以作者又设计了一套reporter系统(Fig.5a)来检测NgAgo插入目的DNA片段的能力。结果表明NgAgo不仅可以准确插入目的基因(Fig.5b),而且也可以进行NHEJ(Fig.5c),且效率不错(Fig.5d)。
- 最后作者对该系统优势总结如下:
1.容错率低,错配一个碱基就明显影响了NgAgo的效率,而错配三个就不起作用了;
2.5'-p-ssDNA在哺乳动物内稀少,这也就决定了胞内不会有gDNA带着NgAgo乱搞;
3.NgAgo一旦与5'-p-ssDNA结合就不会再与其他的5'-p-ssDNA结合;
以上三条都是讲NgAgo脱靶效应低。
4.ssDNAs易于设计和合成,因为它不依赖于PAM序列。这也就是说比较容易推广应用。



 浙公网安备 33010602011771号
浙公网安备 33010602011771号