文献阅读 | Introgressing the Aegilops tauschii genome into wheat as a basis for cereal improvement
Zhou, Y., Bai, S., Li, H. et al. Introgressing the Aegilops tauschii genome into wheat as a basis for cereal improvement. Nat. Plants (2021). https://doi.org/10.1038/s41477-021-00934-w
增加作物产量对于养活世界不断增长的人口是必要的,作物育种者经常利用遗传变异来提高作物产量和质量。然而,小麦 D 基因组狭窄的多样性严重限制了其选择性育种。一个实用的解决方案是通过基因渗入来利用粗山羊草基因组的变异。作者建立了一个快速基因渗入平台,以将粗山羊草中的整体遗传变异转移到小麦中,进而丰富小麦种质库。
作者组装了四个新的参考基因组,对 278 个粗山羊草种质及逆行那个了重测序,建立了粗山羊草的变异全景图(landscape),并通过基因组比较突出了在小麦改良中具有潜在应用的多种功能基因或新型单倍型。此外,作者构建了包含85份材料的粗山羊草核心种质,覆盖了99%以上的遗传变异。其于优良小麦品种杂交,以产生粗山羊草-小麦合成八倍体(A-WSOW)库。实验室和田间试验证实了其在小麦育种方面的巨大潜力。
粗山羊草向小麦的快速渗入
通过将最近的育种和基因组技术集成到快速高通量基因渗入 (RHI) 平台中,作者升级了 SOW 方法。该平台包括以下四个阶段:合成八倍体生成;六倍体化;六倍体稳定化;以及表型和基因分型(图1a)。为确保小麦和粗山羊草同时开花,作者在受控环境室和温室中在快速生长条件下进行了所有直接杂交和基因渗入实验。为了提高 F1形成率,作者使用A. tauschii作为母本与小麦品种杂交。
简而言之,合成八倍体是通过A. tauschii和小麦(通常是改进的目标品种)直接杂交产生四单倍体(ABDD t),然后通过秋水仙碱处理进行胚胎拯救和染色体加倍(图1b)。新产生的合成八倍体(AABBDDDtDt)与目标小麦品种反复回交;这逐渐转移了A. tauschii的变异到六倍体小麦(六倍体化)。进行连续几轮自体受精以稳定具有新变异的渗入小麦品系。最后,使用高通量表型和基因分型平台(图1a)估计和选择A-WIs(A. tauschii-wheat introgression)的种质。
作者可以在 30 个月内完成上述步骤

作者采用中国广泛种植的两个小麦品种周麦18(Z18)和矮抗58作为亲本,创建了一个由85个SOW组成的A. tauschii -wheat SOW(A-WSOW)库,并开发了相应的 A-WI 种群。使用小麦 55 K 单核苷酸多态性 (SNP) 阵列进行的基因分型分析表明,基因渗入等位基因在 Z18 的 D 基因组中均匀分布。A. tauschii特定探针信号的基因组荧光原位杂交 (FISH)还证实了多个等位基因的基因渗入。
此外,数量性状基因座(QTL)连锁分析显示,渗入的等位基因与种群的表型变异显着相关。
系统发育关系和种群结构
鉴于 RHI 平台的有效性和基因组景观在将变异从A. tauschii转移到小麦方面的应用潜力,作者对A. tauschii中可用的种群结构和全球变异进行了全面调查。作者对来自A. tauschii主要分布区(包括中国、西亚和新月沃地东部)的 278 个种质进行了测序(图2a),平均覆盖深度为 13.8 倍。特别是,作者的很大一部分样本,包括 106 个中国种质,没有被任何现有研究覆盖。
全基因组 SNP 数据的系统发育分析确定了两个主要谱系(L1 和 L2),每个谱系由两到三个亚谱系组成(图2b)。
作者的分析证实了早期分析中发现的关系,包括小麦 D 基因组和L2E之间的姐妹关系和最低FST值(图2c),并确定了一个新的亚系 L1EY,它是黄河流域特有的(图2a、b)。作者在此注意到,被多次测序的材料 AL8/78(可能是由于其与小麦 D 基因组的祖先具有潜在的亲和力),被置于 L2W 内(图2b),表明 AL8 /78 不应被视为小麦 D 基因组祖先的近亲。
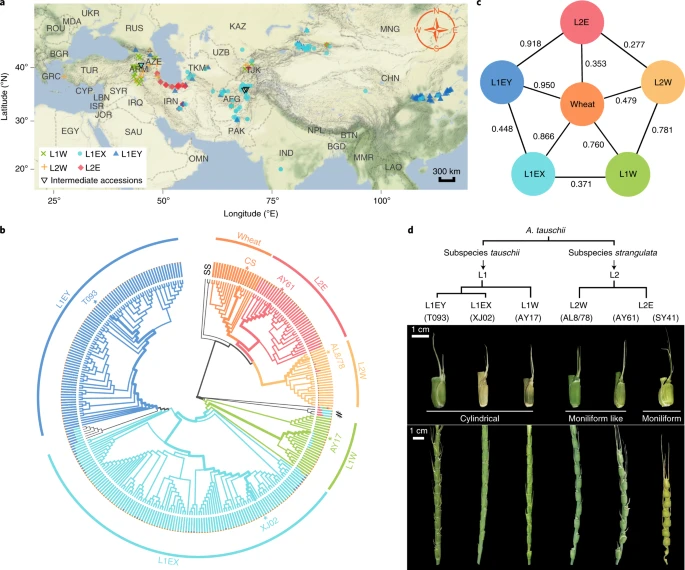
在粗山羊草内,L2的遗传多样性(π)比L1高出两倍(4.3×10-4对2.1×10-4)。遗传多样性从L1 和 L2 的8.0 × 10-4下降到小麦 D 基因组的3.1 × 10-4。如FST值所示,在 L1 和 L2 之间以及 L1 和小麦之间观察到高差异。特别是,黄河流域种质 (L1EY) 与小麦和 L2 的差异最大,表明它们作为补充小麦遗传多样性的未开发资源的价值。
基于新参考基因组的基因组进化
基因组组装和注释
连同来自 L2W 亚系的 AL8/78 的已发布基因组,这些组装不仅将为A. tauschii 的每个主要谱系提供参考基因组,同时也为小麦渗入基因的克隆和功能研究以及更好地了解小麦的起源和进化奠定了基础。
作者将长读序列组装与基因组光学图谱(genome optical mapping)和 Hi-C 支架方法相结合,以产生 AY61 和 T093 的基因组组装。AY61 的总组装基因组大小为 4,222 Mbp,contig N50 和scaffold N50 大小分别为 2.2 和 76.6 Mb。T093 的最终组装基因组大小为 4,121 Mbp。还基于由 T093 基因组引导的长读序列和假染色体锚定获得了 AY17 和 XJ02 的高质量组装。
A. tauschii 的基因组进化
基于 4,461 个单拷贝直向同源基因的系统发育树表明,大约 440 万年前(Ma),A . tauschii进化枝的 D 基因组与 A 基因组进化枝分离(图3a)。A. tauschii 的两个谱系或亚种共享一个共同的 D 祖先,并且可能在大约 2.2 Ma彼此分开(图3a)。作者确定了这些基因组中共线区域的直系同源基因对,然后使用任意两个基因组之间每对直系同源基因之间的同义突变率 ( Ks )估计谱系分歧时间。使用B73和Mo17作为参考,作者发现了两个Ks峰,其中之一对应于亚种差异(Ks = ~0.0009)。作者发现 AY61 和中国春小麦品种 (CS-D) 的 D 基因组在不同对中显示出最高的峰值,这是由于shared orthologous pairs的比例较高且Ks变化很少,这表明它们的关系更密切。另一个峰表明这些 D 基因组可能与共同的 D 祖先发生分歧(Ks = ~0.007),表明A. tauschii基因组的分歧事件晚于玉米(Ks = ~0.025)。
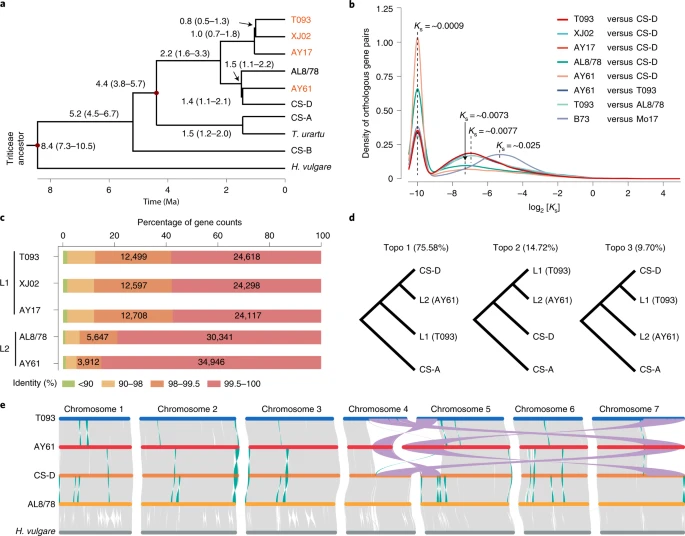
小麦 D 基因组的起源
作者的比较和进化基因组分析证实了 L2E 与小麦 D 基因组群体之间的最密切关系(图2b)。总的来说,作者的全基因组分析支持小麦 D 基因组起源于 L2E 的建议,从 L1 到小麦 D 基因组祖先的基因渗入很少。
A. tauschii 的基因组景观与利用
基因家族的比较基因组学
六个代表性基因组(图4a)由 50,323 个families组成,包括所有六个基因组共享的 20,055 个families、2 到 5 个基因组共享的 18,131 个families(可有可无的基因家族)和 12,137 个亚谱系特异性families(图4b)。可有可无的和亚谱系特异性基因家族占总家族的~60%,并且比核心基因具有显着更高的核苷酸多样性(π)和Ka / Ks<s/ub>。Gene Ontology和 Pfam 注释表明,非生物和生物过程(例如,对压力的反应)和蛋白质稳定性在可有可无的和亚谱系特异性家族中过多。重要的是,CS-D 基因组中不存在 23,580 (47%) 个可有可无的和亚谱系特异性基因家族,再次证实了A. tauschii野生种群作为小麦改良的重要资源的价值。
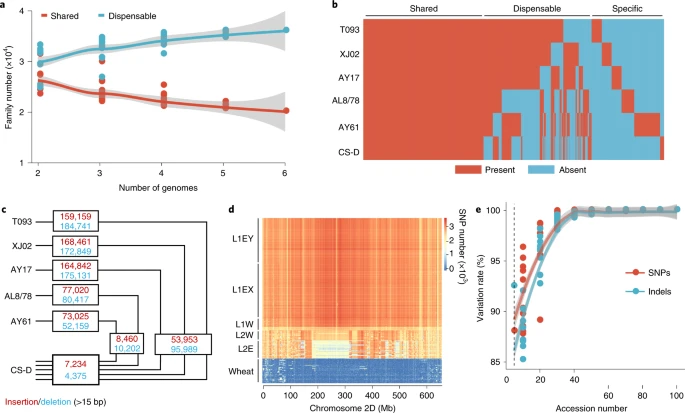
基于代表性参考 D 基因组的基因组变异
作者鉴定了多个SV区域和PAV。中国春基因组中不存在来自5个粗山羊草基因组中共2197个PAV基因。
概述 RHI 用于小麦改良的示例
为了扩大小麦 D 基因组的多样性,作者首先开发了A. tauschii的核心种质。这包括来自所有五个亚系的 85 个种质,涵盖了该物种整体变异的 99% 以上。该核心种质的大小远大于覆盖A. tauschii自然种群整体变异所需的种质数量(图4e)。
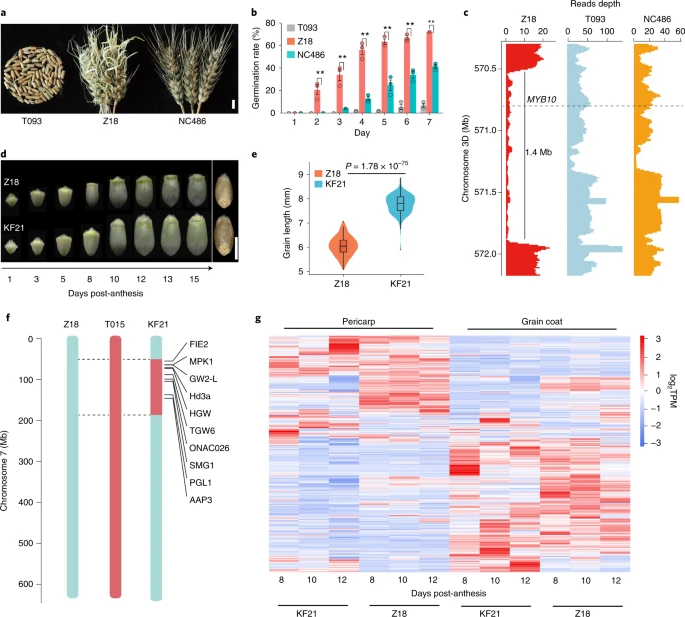
第一个例子是 PHS 抗性(谷物在收获前发芽)。
a, Variation of gene families and shared gene families along with the growing numbers of A. tauschii genomes. Grey shading indicates 95% confidence intervals. b, Presence and absence diagram of gene families in six representative genomes. c, Cladogram depicting a structural variation comparison between the wheat D subgenome of the Chinese Spring wheat cultivar and each of the five representative genomes. We called structural variations by mapping the long-read sequencing data to the wheat D genome of the Chinese Spring wheat cultivar. The numbers of structural variations by genome pairwise comparisons are indicated. Red numbers indicate structural variation lost in the Chinese Spring wheat cultivar, whereas blue numbers indicate structural variation present in the Chinese Spring wheat cultivar. The box-spanning lines represent the shared structural variations between different groups and the wheat D genome of the Chinese Spring wheat cultivar. d, Heatmap representing the density of SNPs that were absent from the wheat D genome population. e, Variation accumulation curves of the wild A. tauschii population. Using the five representative accessions as the starting point (dashed line), the number of variations increased with the number of accessions. Grey shading indicates 95% confidence intervals.
另一个例子与小麦的籽粒大小有关。



 浙公网安备 33010602011771号
浙公网安备 33010602011771号