文献阅读 | Improving and Maintaining Winter Hardiness and Frost Tolerance in Bread Wheat by Genomic Selection
Michel, S., Löschenberger, F., Hellinger, J., Strasser, V., Ametz, C., Pachler, B., ... & Bürstmayr, H. (2019). Improving and maintaining winter hardiness and frost tolerance in bread wheat by genomic selection. Frontiers in plant science, 10, 1195. https://doi.org/10.3389/fpls.2019.01195
作者通过基因组选择(GS)的方法,改善和维持了面包小麦的冬季抗寒性和抗冻性。
背景
小麦的抗寒性是一个复杂的性状,它受多种环境因素的强烈影响,例如是否存在积雪、土壤肥力、土壤起伏或覆冰,以及诸如疾病压力或虫害等生物因素。除间接影响外,对冰点以下的温度的耐受性通常对于整个冬季的生存至关重要。不同基因型对暴露于寒冷之中的能力上有所差异,同时这一能力也与特定的春化要求、田间可能发生的重复解冻-冷冻循环的差异反应有关。
由于田间环境下土壤自身差异、积雪量差异等因素,选择时对候选者的能力估算会有所偏差。野外试验的替代方案是在雪掩体中进行半控制实验,或在具有预定温度程序的气候箱中进行控制霜冻耐受性的实验。后一种方法包括评估50%的植物因冷冻而死亡的致死温度(LT50)、在−5°C较温和的冻结温度下长期储存后进行恢复,或将植物短时间暴露在严重的冻结温度下,并在预先确定的温度下测量霜冻百分率。以这种方式对冬小麦进行的测试通常包括一段时间的冷硬化以诱导耐寒性的生理机制,如水溶性碳水化合物的积累。然而,这种在气候室中进行的抗冻性试验费时费力,费用昂贵,而且通常仅限于少数基因型,使得基因组育种方法成为应用育种计划的一个有吸引力的替代方法。
因此,进行了对小麦抗寒性和抗冻性的遗传结构进行了大量研究,揭示了位于5号染色体的同源位点Vrn-A1、Vrn-B1和Vrn-D1对小麦春化反应和抗冻性的重要性。后两个基因座至少在欧洲冬小麦中是冬型等位基因的固定位点,尽管它们对兼性小麦品种的培育很有意义。另一方面,Vrn-A1的拷贝数变异影响冬小麦的春化反应和抗冻性。尽管已经有1-3个Vrn-A1拷贝的小麦品系,但其拷贝数的变异只能解释欧洲冬小麦品系中2.9%的抗寒性遗传变异。
除了Vrn基因座的多效性外,5B染色体上的Fr-2B基因座也被报道与抗冻性有关。在这种情况下,对低温胁迫的耐受性降低是由于基因座上11个基因的缺失而导致的功能丧失,尽管这在欧洲冬小麦中非常罕见(Fr-2B对霜冻敏感的等位基因频率低于2%)。另一方面,5A染色体上Fr-A2耐霜冻位点的等位基因频率更为平衡,其对硬粒小麦和冬小麦的耐霜冻性和抗寒性的影响更大,是一个值得标记辅助选择的目标。Fr-A2在Vrn-A1附近30-46cM处的图谱位置已经确定,而该位点本身由一个编码多个C-重复结合因子(CBF)的基因簇组成。这些转录激活蛋白的拷贝数变异在调控冷驯化和抗冻性的相关途径中起着重要作用,特别是CBF-A14的拷贝数变异与抗冻性和抗寒性密切相关。然而,CBF-A14的多等位性不能用双等位SNP标记来充分描述(双等位SNP标记通常用于应用植物育种项目的指纹分析)。因此,有人建议通过与Fr-A2相关的两个SNP标记的单倍体来捕获拷贝数变异,这解释面包小麦了高达24%的抗寒性遗传变异。
尽管如此,Fr-A2只能解释基因变异的一部分,从而限制了基因组育种计划中对选择的可实现反应,除少数几个主要QTL外,据报道还有许多次要QTL影响霜冻耐受性和抗寒性。利用全基因组分布标记的基因组预测方法可以更有效地定位这些位点,其中表型和基因型训练群体通过模拟两个群体之间的遗传关系来预测验证/选择群体中个体的表现。利用这样的基因组预测进行选择已经在一些国家和国际小麦育种项目中得到常规实施,这是因为利用新的指纹技术(如浅测序)降低了基因分型成本。浅测序或者在靶向基因分型测序(GBS)以及定制和优化的SNP阵列上使用预选的标记集。基因分型的这些进展使得对多种农艺性状进行早代标记辅助和基因组选择成为可能,这对于困难、费力和昂贵的表型性状(如抗寒性或抗冻性)特别有趣。因此,本研究的目的是1)比较标记辅助基因组选择在抗寒性和抗冻性方面的优点,2)评估利用高密度标记图谱或减少的预选标记集将这两个性状与基因组选择指数相结合的潜力。
在对表型数据的处理中,使用了一个线性模型进行分析。其中,对BLUE和遗传力进行了估计。
基因型数据采用了DarT GBS方法进行获得,并使用了missForest算法对缺失的数据点进行估计。
GS
标记辅助和基因组预测环节,作者首先通过100次重复采样方案评估了冬季抗寒性和耐霜冻性的基因组预测优点,该方案采用了2012年的实地数据,并分别与2017年气候室试验中测得的耐霜冻性进行了评估。作者首先计算了亲缘关系矩阵\(K\),随后通过包括获得的基因组关系矩阵的最佳线性基因组预测(GBLUP)模型,得出基因组估计育种值(GEBV)\(y\)。考虑到Fr-A2基因座对冬季耐寒性和耐霜冻性均具有较大影响的先验知识,作者在单独的加权基因组最佳线性无偏预测(WBLUP)模型中以固定效应建模的方式进一步更新了y的计算模型。
除了通过整合Fr-A2基因座上的拷贝数变异来测试基因组预测的优点及其增强功能外,还需要进一步研究获得一组减少的标记的可能性,这些标记将在很大程度上保持先前设计定制SNP芯片或靶向GBS的模型的预测准确性。为此,作者给出了该场景下的基于线性混合模型。
最后,作者引入了岭回归最佳线性无偏预测(RR-BLUP)模型,旨在寻找可能影响冬季的耐寒性或抗冻性的、覆盖整个基因组的标记-性状关联。上述模型可以调用R中的软件包。
评估
首先通过在2012年冬季耐寒性和2017年耐霜冻性测试的亚群体中使用重采样方案评估基因组预测的优点。在两个数据集中随机选择21个标记后可发现,基因组选择的预测准确率明显高于标记辅助选择(下图)。
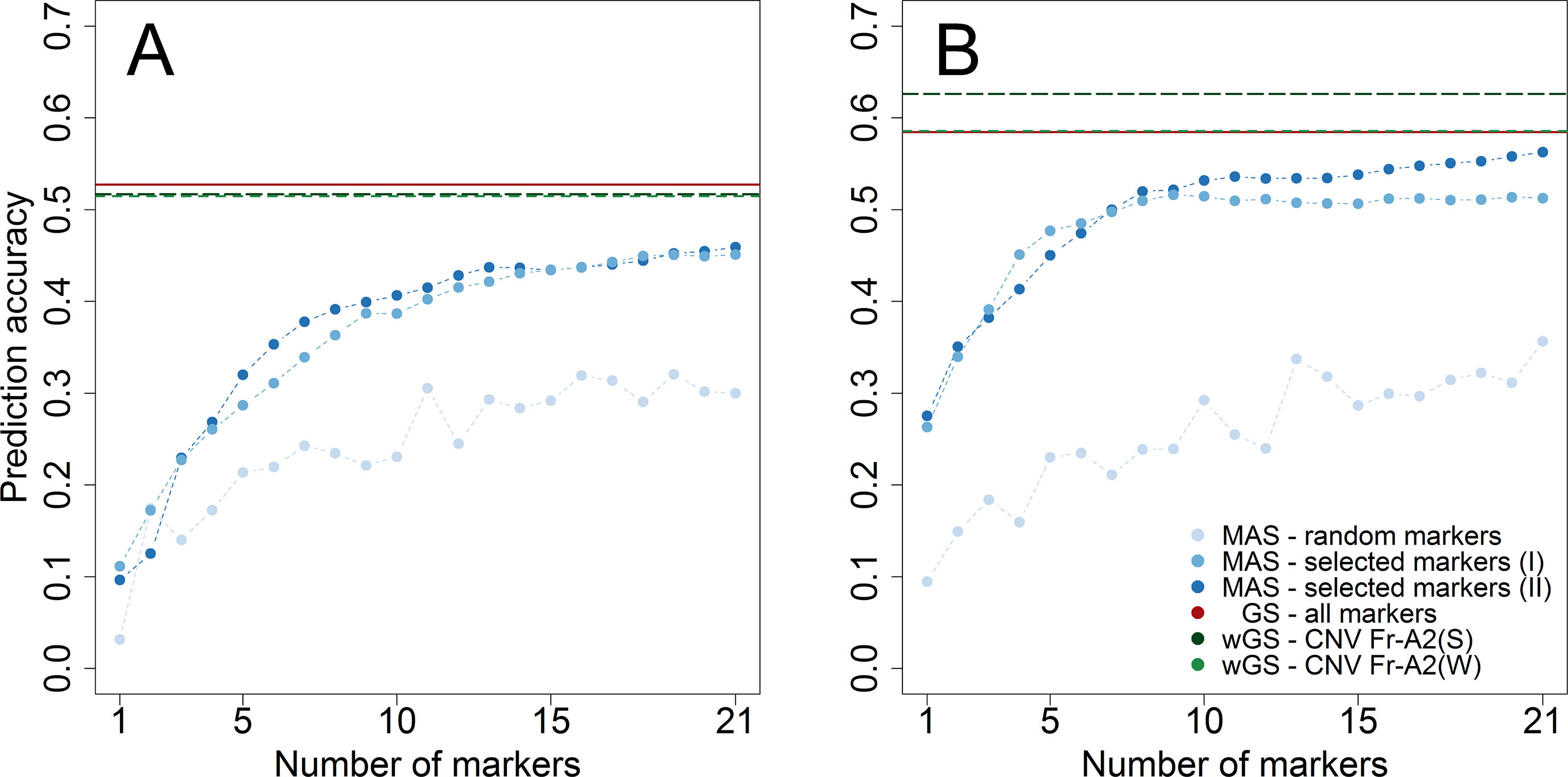
尽管如此,通过预选21个标记(rMAS=0.459)的标记辅助预测,可以获得几乎相同水平的耐寒性预测准确率,这与采用一千多个标记(rGS=0.527)的基因组预测一致。
对于抗冻性的预测也可以进行类似的发现,其中用预选标记辅助预测的效果仅比基因组预测(rGS=0.585)稍差(rMAS=0.563)。后者可以通过将Fr-A2基因座的拷贝数变异与单倍体CNV Fr-A2(S)作为固定效应(rwGS=0.626)相关联的模型来进一步改进。



 浙公网安备 33010602011771号
浙公网安备 33010602011771号