文献阅读 | Optimal Designs for Genomic Selection in Hybrid Crops
Guo T, Yu X, Li X, et al. Optimal designs for genomic selection in hybrid crops[J]. Molecular plant, 2019, 12(3): 390-401.
在给定的资源下,通过基因组学方法从众多潜在组合中鉴定出优良的杂种是一项长期挑战。本文通过基于聚类(clustering),图形网络分析(graphic network analysis)和遗传交配方案(genetic mating scheme)的方法建立训练样本集,进行从基因型向表型的预测,并得出结论:使用整个集合中2%-13%的样本即可构建一个有效(effective)的预测模型。通过比较不同的训练集设计方法,我们表明代表性的子集选择方法的效果优于随机抽样,并且MaxCD,PAM和FURS的效果优于PEVmean和CDmean。另一方面,在不同的物种特征组合中,不同的方法要优于其他方法。
这项研究的发现首先是通过对玉米进行的经验性研究发现的,然后通过对种间杂种的分析以及之前在小麦和水稻中发布的两个数据集的分析得到证实。在这项研究中,作者调查了使用三种物种的小型杂种集来预测所有杂种大基因型空间的表现。作者使用的百分比是通常数据挖掘研究所建议的15%(Han et al., 2011)。作者检查的百分比上限为32%,这提供了比较的背景。
针对自交群体的训练集设计的研究(Rincent et al., 2012, Akdemir et al., 2015, Isidro et al., 2015, Lorenz and Smith, 2015, Marulanda et al., 2015)显示出令人鼓舞的结果。这些研究集中于从混合模型方程式得出的两个参数,即最小化预测误差方差的平均值(PEVmean)或最大化确定系数的平均值(CDmean),并使用了穷举搜索或遗传算法。
杂交种的基因组预测和训练种群设计与自交系不同。首先,在为近交系生成基因组估计育种值的同时,使用包含加性和优势基因组关系矩阵的协方差矩阵为杂种生成预测的基因型值。其次,除了亲本近亲之间的遗传关系外,在杂交过程中还引入了半同胞关系。第三,不像近交群体,杂交群体中只有训练集和基于预测值被选出的个体需要采收(derived)和测表型。最后,省去了对不好的杂交种进行收获(obtaining)和测表型的过程,突出了设计在杂交种基因组预测中的额外重要性,其数量是亲本自交系的函数。
在这项研究中,作者检查了可用于自交系的杂种表现预测的训练集设计,这些自交系构成了需要选择杂种组合的整体遗传空间。对于具有确定的杂种模式的杂交作物,例如玉米,黑麦和高粱,潜在杂交种的数量是来自不同杂种组的近交系数量的乘积(\(n_1 \times n_2\),其中\(n_1\)是其中一组的数量,\(n_2\)是另一组)。对于需要开发杂交模式的农作物,例如小麦,它是自交数(n)的一元二次方程(\(\frac{n(n-1)}{2}\))。
作者设计并测试了三种方法来抽取代表性子集,以建立用于杂交种中基因组预测的训练集。

对于小麦这一仍处于杂交育种早期的作物,作者使用了(Zhao et al., 2015) 的文章中由72个适应于欧洲中部(Central Europe)构成的2556个杂交小麦数据集进行测试。这些自交系已被分成两组,分别属于两个杂种群体的各36个品系。基于基因型数据的PCA图表明,这两组之间的分离不强(图4 A和4B),这与缺乏密集的发散选择(the lack of intensive divergent selection)相符。通过分级聚类对近交种进行排序后,从基因组关系矩阵中出现了可检测的分组模式(图4 C)。从表型上看,组间杂种的平均籽粒产量为10.833 Mg ha -1,略高于两组中每组的杂交平均数(10.827Mg/ha和10.670Mg/ha)(图4 D),总体上与基因组关系矩阵的预期一致。
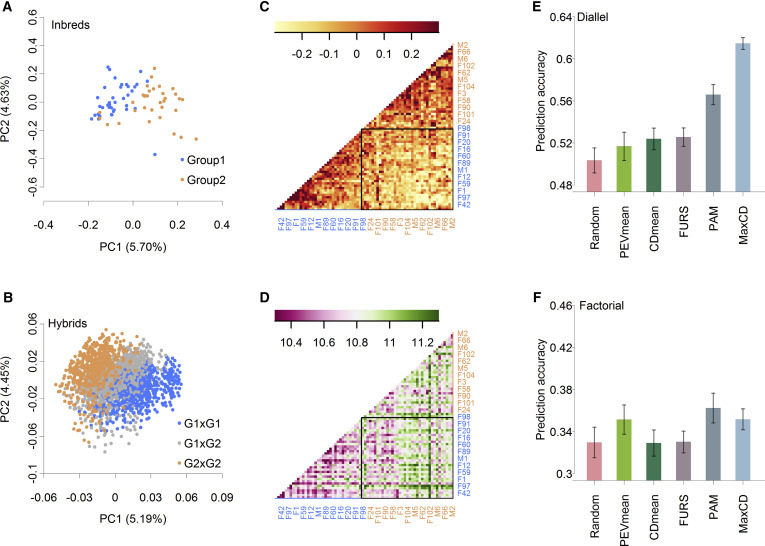
在每次运行中,杂种总数为1770,每种方法(包括随机抽样)生成了一个训练集,其大小为所有杂种的2.5%。与随机抽样相比,每种方法的代表性子集产生的预测准确性值更高(图4 E)。MaxCD的值增加了18%,PAM的值增加了11%,FURS的值增加了4%。与随机采样相比,这些方法的预测精度方差更低,这是理想的。尽管PEVmean,CDmean和FURS的准确度要高于随机抽样,但P值> 0.01表示无统计学差异。在训练百分比分析中,在较低的样本量末端观察到了PAM和FURS的优势(补充图5)。



 浙公网安备 33010602011771号
浙公网安备 33010602011771号