t-SNE完整笔记
http://www.datakit.cn/blog/2017/02/05/t_sne_full.html
t-SNE(t-distributed stochastic neighbor embedding)是用于降维的一种机器学习算法,是由 Laurens van der Maaten 和 Geoffrey Hinton在08年提出来。此外,t-SNE 是一种非线性降维算法,非常适用于高维数据降维到2维或者3维,进行可视化。
t-SNE是由SNE(Stochastic Neighbor Embedding, SNE; Hinton and Roweis, 2002)发展而来。我们先介绍SNE的基本原理,之后再扩展到t-SNE。最后再看一下t-SNE的实现以及一些优化。
目录
1.SNE
1.1基本原理
SNE是通过仿射(affinitie)变换将数据点映射到概率分布上,主要包括两个步骤:
- SNE构建一个高维对象之间的概率分布,使得相似的对象有更高的概率被选择,而不相似的对象有较低的概率被选择。
- SNE在低维空间里在构建这些点的概率分布,使得这两个概率分布之间尽可能的相似。
我们看到t-SNE模型是非监督的降维,他跟kmeans等不同,他不能通过训练得到一些东西之后再用于其它数据(比如kmeans可以通过训练得到k个点,再用于其它数据集,而t-SNE只能单独的对数据做操作,也就是说他只有fit_transform,而没有fit操作)
1.2 SNE原理推导
SNE是先将欧几里得距离转换为条件概率来表达点与点之间的相似度。具体来说,给定一个N个高维的数据 x1,...,xNx1,...,xN(注意N不是维度), t-SNE首先是计算概率pijpij,正比于xixi和xjxj之间的相似度(这种概率是我们自主构建的),即:
这里的有一个参数是σiσi,对于不同的点xixi取值不一样,后续会讨论如何设置。此外设置px∣x=0px∣x=0,因为我们关注的是两两之间的相似度。
那对于低维度下的yiyi,我们可以指定高斯分布为方差为12√12,因此它们之间的相似度如下:
同样,设定qi∣i=0qi∣i=0.
如果降维的效果比较好,局部特征保留完整,那么 pi∣j=qi∣jpi∣j=qi∣j, 因此我们优化两个分布之间的距离-KL散度(Kullback-Leibler divergences),那么目标函数(cost function)如下:
这里的PiPi表示了给定点xixi下,其他所有数据点的条件概率分布。需要注意的是KL散度具有不对称性,在低维映射中不同的距离对应的惩罚权重是不同的,具体来说:距离较远的两个点来表达距离较近的两个点会产生更大的cost,相反,用较近的两个点来表达较远的两个点产生的cost相对较小(注意:类似于回归容易受异常值影响,但效果相反)。即用较小的 qj∣i=0.2qj∣i=0.2 来建模较大的 pj∣i=0.8pj∣i=0.8, cost=plog(pq)plog(pq)=1.11,同样用较大的qj∣i=0.8qj∣i=0.8来建模较大的pj∣i=0.2pj∣i=0.2, cost=-0.277, 因此,SNE会倾向于保留数据中的局部特征。
思考:了解了基本思路之后,你会怎么选择σσ,固定初始化?
下面我们开始正式的推导SNE。首先不同的点具有不同的σiσi,PiPi的熵(entropy)会随着σiσi的增加而增加。SNE使用困惑度(perplexity)的概念,用二分搜索的方式来寻找一个最佳的σσ。其中困惑度指:
这里的H(Pi)H(Pi)是PiPi的熵,即:
困惑度可以解释为一个点附近的有效近邻点个数。SNE对困惑度的调整比较有鲁棒性,通常选择5-50之间,给定之后,使用二分搜索的方式寻找合适的σσ
那么核心问题是如何求解梯度了,目标函数等价于∑∑−plog(q)∑∑−plog(q)这个式子与softmax非常的类似,我们知道softmax的目标函数是∑−ylogp∑−ylogp,对应的梯度是y−py−p(注:这里的softmax中y表示label,p表示预估值)。 同样我们可以推导SNE的目标函数中的i在j下的条件概率情况的梯度是2(pi∣j−qi∣j)(yi−yj)2(pi∣j−qi∣j)(yi−yj), 同样j在i下的条件概率的梯度是2(pj∣i−qj∣i)(yi−yj)2(pj∣i−qj∣i)(yi−yj), 最后得到完整的梯度公式如下:
在初始化中,可以用较小的σσ下的高斯分布来进行初始化。为了加速优化过程和避免陷入局部最优解,梯度中需要使用一个相对较大的动量(momentum)。即参数更新中除了当前的梯度,还要引入之前的梯度累加的指数衰减项,如下:
这里的Y(t)Y(t)表示迭代t次的解,ηη表示学习速率,α(t)α(t)表示迭代t次的动量。
此外,在初始优化的阶段,每次迭代中可以引入一些高斯噪声,之后像模拟退火一样逐渐减小该噪声,可以用来避免陷入局部最优解。因此,SNE在选择高斯噪声,以及学习速率,什么时候开始衰减,动量选择等等超参数上,需要跑多次优化才可以。
思考:SNE有哪些不足? 面对SNE的不足,你会做什么改进?
2.t-SNE
尽管SNE提供了很好的可视化方法,但是他很难优化,而且存在”crowding problem”(拥挤问题)。后续中,Hinton等人又提出了t-SNE的方法。与SNE不同,主要如下:
- 使用对称版的SNE,简化梯度公式
- 低维空间下,使用t分布替代高斯分布表达两点之间的相似度
t-SNE在低维空间下使用更重长尾分布的t分布来避免crowding问题和优化问题。在这里,首先介绍一下对称版的SNE,之后介绍crowding问题,之后再介绍t-SNE。
2.1 Symmetric SNE
优化pi∣jpi∣j和qi∣jqi∣j的KL散度的一种替换思路是,使用联合概率分布来替换条件概率分布,即P是高维空间里各个点的联合概率分布,Q是低维空间下的,目标函数为:
这里的piipii,qiiqii为0,我们将这种SNE称之为symmetric SNE(对称SNE),因为他假设了对于任意i,pij=pji,qij=qjipij=pji,qij=qji,因此概率分布可以改写为:
这种表达方式,使得整体简洁了很多。但是会引入异常值的问题。比如xixi是异常值,那么∣∣xi−xj∣∣2∣∣xi−xj∣∣2会很大,对应的所有的j, pijpij都会很小(之前是仅在xixi下很小),导致低维映射下的yiyi对cost影响很小。
思考: 对于异常值,你会做什么改进?pipi表示什么?
为了解决这个问题,我们将联合概率分布定义修正为: pij=pi∣j+pj∣i2pij=pi∣j+pj∣i2, 这保证了∑jpij>12n∑jpij>12n, 使得每个点对于cost都会有一定的贡献。对称SNE的最大优点是梯度计算变得简单了,如下:
实验中,发现对称SNE能够产生和SNE一样好的结果,有时甚至略好一点。
2.2 Crowding问题
拥挤问题就是说各个簇聚集在一起,无法区分。比如有一种情况,高维度数据在降维到10维下,可以有很好的表达,但是降维到两维后无法得到可信映射,比如降维如10维中有11个点之间两两等距离的,在二维下就无法得到可信的映射结果(最多3个点)。 进一步的说明,假设一个以数据点xixi为中心,半径为r的m维球(三维空间就是球),其体积是按rmrm增长的,假设数据点是在m维球中均匀分布的,我们来看看其他数据点与xixi的距离随维度增大而产生的变化。
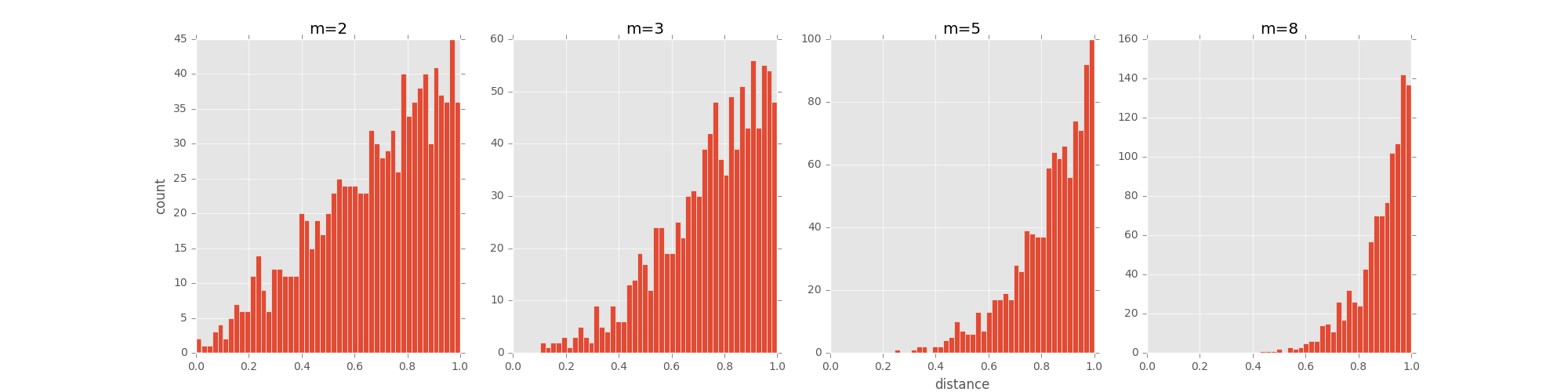
从上图可以看到,随着维度的增大,大部分数据点都聚集在m维球的表面附近,与点xixi的距离分布极不均衡。如果直接将这种距离关系保留到低维,就会出现拥挤问题。
怎么解决crowding问题呢?
Cook et al.(2007) 提出一种slight repulsion的方式,在基线概率分布(uniform background)中引入一个较小的混合因子ρρ,这样qijqij就永远不会小于2ρn(n−1)2ρn(n−1) (因为一共了n(n-1)个pairs),这样在高维空间中比较远的两个点之间的qijqij总是会比pijpij大一点。这种称之为UNI-SNE,效果通常比标准的SNE要好。优化UNI-SNE的方法是先让ρρ为0,使用标准的SNE优化,之后用模拟退火的方法的时候,再慢慢增加ρρ. 直接优化UNI-SNE是不行的(即一开始ρρ不为0),因为距离较远的两个点基本是一样的qijqij(等于基线分布), 即使pijpij很大,一些距离变化很难在qijqij中产生作用。也就是说优化中刚开始距离较远的两个聚类点,后续就无法再把他们拉近了。
2.3 t-SNE
对称SNE实际上在高维度下 另外一种减轻”拥挤问题”的方法:在高维空间下,在高维空间下我们使用高斯分布将距离转换为概率分布,在低维空间下,我们使用更加偏重长尾分布的方式来将距离转换为概率分布,使得高维度下中低等的距离在映射后能够有一个较大的距离。
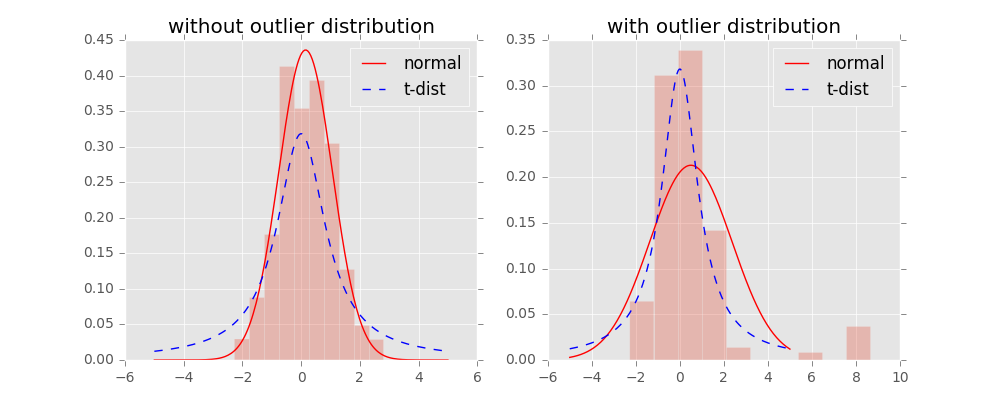
我们对比一下高斯分布和t分布(如上图,code见probability/distribution.md), t分布受异常值影响更小,拟合结果更为合理,较好的捕获了数据的整体特征。
使用了t分布之后的q变化,如下:
此外,t分布是无限多个高斯分布的叠加,计算上不是指数的,会方便很多。优化的梯度如下:
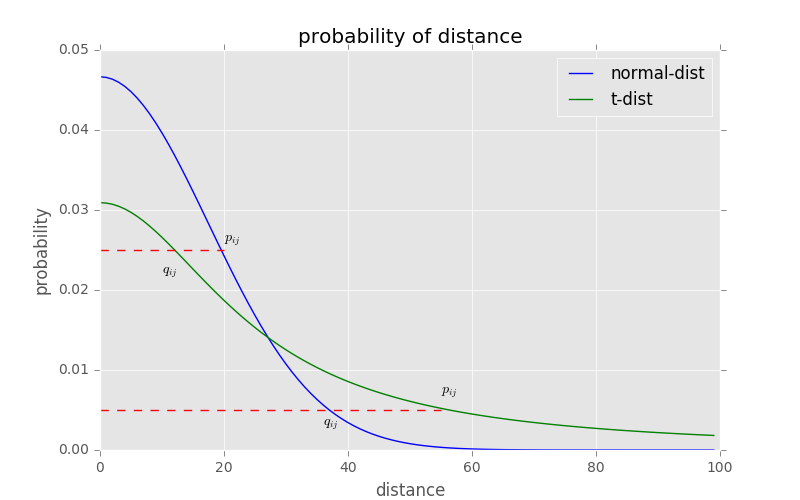
t-sne的有效性,也可以从上图中看到:横轴表示距离,纵轴表示相似度, 可以看到,对于较大相似度的点,t分布在低维空间中的距离需要稍小一点;而对于低相似度的点,t分布在低维空间中的距离需要更远。这恰好满足了我们的需求,即同一簇内的点(距离较近)聚合的更紧密,不同簇之间的点(距离较远)更加疏远。
总结一下,t-SNE的梯度更新有两大优势:
- 对于不相似的点,用一个较小的距离会产生较大的梯度来让这些点排斥开来。
- 这种排斥又不会无限大(梯度中分母),避免不相似的点距离太远。
2.4 算法过程
算法详细过程如下:
- Data: X=x1,...,xnX=x1,...,xn
- 计算cost function的参数:困惑度Perp
- 优化参数: 设置迭代次数T, 学习速率ηη, 动量α(t)α(t)
- 目标结果是低维数据表示 YT=y1,...,ynYT=y1,...,yn
- 开始优化
- 计算在给定Perp下的条件概率pj∣ipj∣i(参见上面公式)
- 令 pij=pj∣i+pi∣j2npij=pj∣i+pi∣j2n
- 用 N(0,10−4I)N(0,10−4I) 随机初始化 Y
- 迭代,从 t = 1 到 T, 做如下操作:
- 计算低维度下的 qijqij(参见上面的公式)
- 计算梯度(参见上面的公式)
- 更新 Yt=Yt−1+ηdCdY+α(t)(Yt−1−Yt−2)Yt=Yt−1+ηdCdY+α(t)(Yt−1−Yt−2)
- 结束
- 结束
优化过程中可以尝试的两个trick:
- 提前压缩(early compression):开始初始化的时候,各个点要离得近一点。这样小的距离,方便各个聚类中心的移动。可以通过引入L2正则项(距离的平方和)来实现。
- 提前夸大(early exaggeration):在开始优化阶段,pijpij乘以一个大于1的数进行扩大,来避免因为qijqij太小导致优化太慢的问题。比如前50次迭代,pijpij乘以4
优化的过程动态图如下:
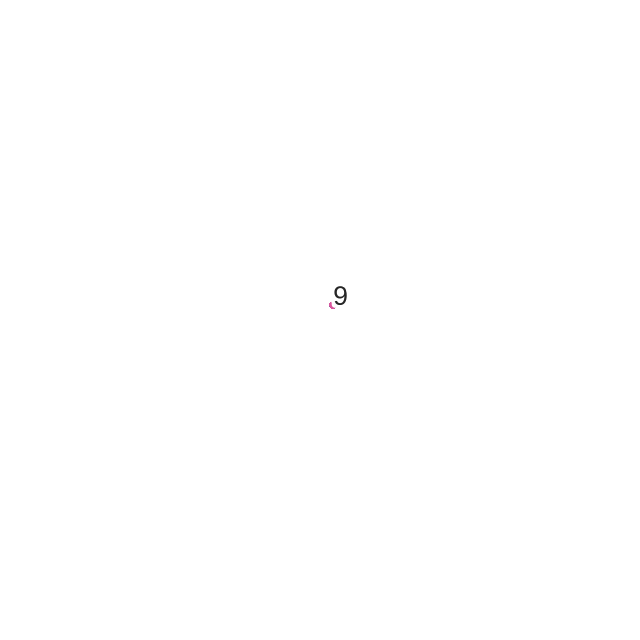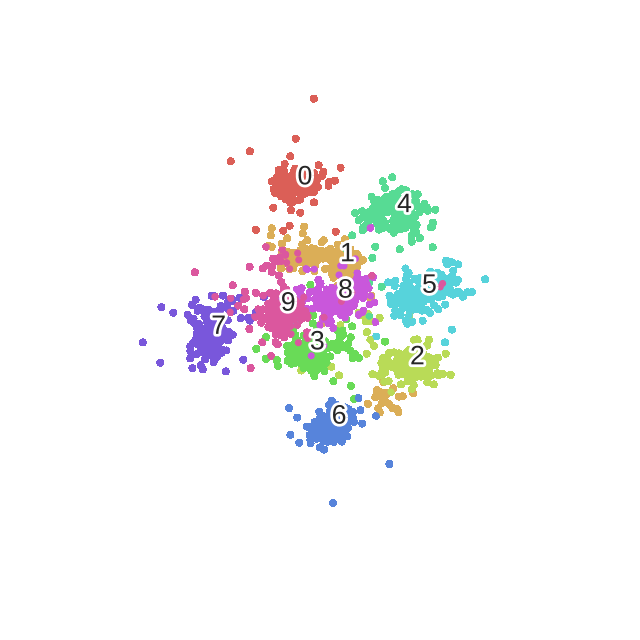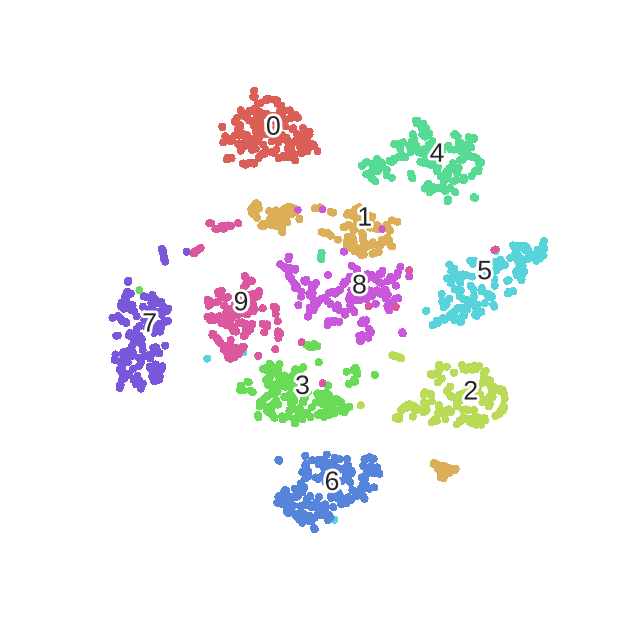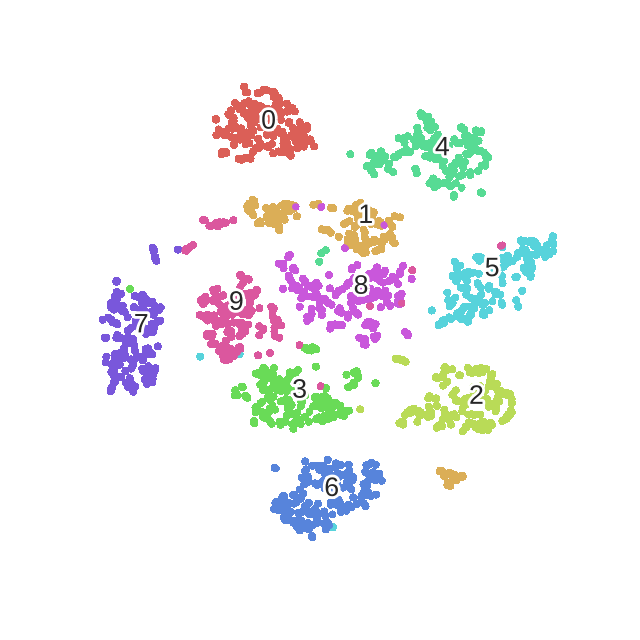
2.5 不足
主要不足有四个:
- 主要用于可视化,很难用于其他目的。比如测试集合降维,因为他没有显式的预估部分,不能在测试集合直接降维;比如降维到10维,因为t分布偏重长尾,1个自由度的t分布很难保存好局部特征,可能需要设置成更高的自由度。
- t-SNE倾向于保存局部特征,对于本征维数(intrinsic dimensionality)本身就很高的数据集,是不可能完整的映射到2-3维的空间
- t-SNE没有唯一最优解,且没有预估部分。如果想要做预估,可以考虑降维之后,再构建一个回归方程之类的模型去做。但是要注意,t-sne中距离本身是没有意义,都是概率分布问题。
- 训练太慢。有很多基于树的算法在t-sne上做一些改进
3.变种
后续有机会补充。
- multiple maps of t-SNE
- parametric t-SNE
- Visualizing Large-scale and High-dimensional Data
4.参考文档
- Maaten, L., & Hinton, G. (2008). Visualizing data using t-SNE. Journal of Machine Learning Research.
5. 代码
文中的插图绘制:
# coding:utf-8
import numpy as np
from numpy.linalg import norm
from matplotlib import pyplot as plt
plt.style.use('ggplot')
def sne_crowding():
npoints = 1000 # 抽取1000个m维球内均匀分布的点
plt.figure(figsize=(20, 5))
for i, m in enumerate((2, 3, 5, 8)):
# 这里模拟m维球中的均匀分布用到了拒绝采样,
# 即先生成m维立方中的均匀分布,再剔除m维球外部的点
accepts = []
while len(accepts) < 1000:
points = np.random.rand(500, m)
accepts.extend([d for d in norm(points, axis=1)
if d <= 1.0]) # 拒绝采样
accepts = accepts[:npoints]
ax = plt.subplot(1, 4, i+1)
if i == 0:
ax.set_ylabel('count')
if i == 2:
ax.set_xlabel('distance')
ax.hist(accepts, bins=np.linspace(0., 1., 50))
ax.set_title('m=%s' %m)
plt.savefig("./images/sne_crowding.png")
x = np.linspace(0, 4, 100)
ta = 1 / (1 + np.square(x))
tb = np.sum(ta) - 1
qa = np.exp(-np.square(x))
qb = np.sum(qa) - 1
def sne_norm_t_dist_cost():
plt
