scikit-learning教程(二)统计学习科学数据处理的教程二
模型选择:选择估计量及其参数
得分和交叉验证的分数
如我们所看到的,每个估计者都会公开一种score可以判断新数据的拟合质量(或预测)的方法。越大越好。
>>> from sklearn import datasets, svm
>>> digits = datasets.load_digits()
>>> X_digits = digits.data
>>> y_digits = digits.target
>>> svc = svm.SVC(C=1, kernel='linear')
>>> svc.fit(X_digits[:-100], y_digits[:-100]).score(X_digits[-100:], y_digits[-100:])
0.97999999999999998
为了更好地测量预测精度(我们可以将其用作模型的拟合优势),我们可以连续地将数据分割成用于训练和测试的折叠:
>>> import numpy as np
>>> X_folds = np.array_split(X_digits, 3)
>>> y_folds = np.array_split(y_digits, 3)
>>> scores = list()
>>> for k in range(3):
... # We use 'list' to copy, in order to 'pop' later on
... X_train = list(X_folds)
... X_test = X_train.pop(k)
... X_train = np.concatenate(X_train)
... y_train = list(y_folds)
... y_test = y_train.pop(k)
... y_train = np.concatenate(y_train)
... scores.append(svc.fit(X_train, y_train).score(X_test, y_test))
>>> print(scores)
[0.93489148580968284, 0.95659432387312182, 0.93989983305509184]
这被称为KFold交叉验证。
交叉验证生成器
Scikit学习有一系列课程,可用于生成流行交叉验证策略的列车/测试指标列表。
它们公开了split一种接受要分割的输入数据集的方法,并为所选交叉验证策略的每次迭代产生列车/测试集合索引。
此示例显示了该split方法的示例用法。
>>> from sklearn.model_selection import KFold, cross_val_score
>>> X = ["a", "a", "b", "c", "c", "c"]
>>> k_fold = KFold(n_splits=3)
>>> for train_indices, test_indices in k_fold.split(X):
... print('Train: %s | test: %s' % (train_indices, test_indices))
Train: [2 3 4 5] | test: [0 1]
Train: [0 1 4 5] | test: [2 3]
Train: [0 1 2 3] | test: [4 5]
然后可以轻松执行交叉验证:
>>> kfold = KFold(n_splits=3)
>>> [svc.fit(X_digits[train], y_digits[train]).score(X_digits[test], y_digits[test])
... for train, test in k_fold.split(X_digits)]
[0.93489148580968284, 0.95659432387312182, 0.93989983305509184]
交叉验证分数可以使用cross_val_score助手直接计算 。给定估计器,交叉验证对象和输入数据集,cross_val_score将数据重复分为训练和测试集,使用训练集训练估计器,并根据每次交叉验证迭代的测试集计算得分。
默认情况下,使用估计器的score方法来计算单个分数。
参考指标模块,了解更多可用评分方法。
>>> cross_val_score(svc, X_digits, y_digits, cv=k_fold, n_jobs=-1)
array([ 0.93489149, 0.95659432, 0.93989983])
n_jobs = -1表示将在计算机的所有CPU上分派计算。
或者,scoring可以提供参数以指定替代评分方法。
>>> cross_val_score(svc, X_digits, y_digits, cv=k_fold, ... scoring='precision_macro') array([ 0.93969761, 0.95911415, 0.94041254])交叉验证生成器
KFold (n_splits,shuffle,random_state) |
StratifiedKFold (n_iter,test_size,train_size,random_state) |
GroupKFold (n_splits,shuffle,random_state) |
| 将其分为K个折叠,K-1上的火车,然后在左侧测试。 | 与K-Fold相同,但保留每个折叠内的类分布。 | 确保同一组不在测试和训练集中。 |
ShuffleSplit (n_iter,test_size,train_size,random_state) |
StratifiedShuffleSplit |
GroupShuffleSplit |
| 根据随机排列产生列车/测试指标。 | 与shuffle split相同,但保留了每次迭代中的类分布。 | 确保同一组不在测试和训练集中。 |
LeaveOneGroupOut () |
LeavePGroupsOut (p)的 |
LeaveOneOut () |
| 使用组数组来对观察进行分组。 | 离开P组。 | 留下一个观察。 |
LeavePOut (p)的 |
PredefinedSplit |
| 留下P观察。 | 根据预定义的分割生成火车/测试指标。 |
行使

在数字数据集上,绘制SVC 具有线性内核的估计器的交叉验证分数作为参数的函数C(使用点对数网格,从1到10)。
import numpy as np
from sklearn.model_selection import cross_val_score
from sklearn import datasets, svm
digits = datasets.load_digits()
X = digits.data
y = digits.target
svc = svm.SVC(kernel='linear')
C_s = np.logspace(-10, 0, 10)
解决方案: 数字数据集练习的交叉验证
网格搜索和交叉验证的估计
网格搜索
scikit-learn提供了一个对象,给定的数据在参数网格中的估计器拟合期间计算分数,并选择参数以最大化交叉验证分数。该对象在构建期间使用估计器,并公开估计器API:
>>> from sklearn.model_selection import GridSearchCV, cross_val_score
>>> Cs = np.logspace(-6, -1, 10)
>>> clf = GridSearchCV(estimator=svc, param_grid=dict(C=Cs),
... n_jobs=-1)
>>> clf.fit(X_digits[:1000], y_digits[:1000])
GridSearchCV(cv=None,...
>>> clf.best_score_
0.925...
>>> clf.best_estimator_.C
0.0077...
>>> # Prediction performance on test set is not as good as on train set
>>> clf.score(X_digits[1000:], y_digits[1000:])
0.943...
默认情况下,GridSearchCV使用三重交叉验证。然而,如果它检测到分类器被传递,而不是一个回归,它将使用分层3倍。
嵌套交叉验证
>>> cross_val_score(clf, X_digits, y_digits)
...
array([ 0.938..., 0.963..., 0.944...])
并行执行两个交叉验证循环:一个通过 GridSearchCV估计器进行设置gamma,另一个通过 cross_val_score测量估计器的预测性能。得出的分数是对新数据的预测分数的无偏估计。
警告
您不能使用并行计算来嵌套对象(n_jobs不同于1)。
交叉验证的估计
可以在逐个算法的基础上更有效地进行交叉验证来设置参数。这就是为什么,对于某些估计,scikit学习公开交叉验证:评估通过交叉验证自动设置其参数的估计器性能估计器:
>>> from sklearn import linear_model, datasets
>>> lasso = linear_model.LassoCV()
>>> diabetes = datasets.load_diabetes()
>>> X_diabetes = diabetes.data
>>> y_diabetes = diabetes.target
>>> lasso.fit(X_diabetes, y_diabetes)
LassoCV(alphas=None, copy_X=True, cv=None, eps=0.001, fit_intercept=True,
max_iter=1000, n_alphas=100, n_jobs=1, normalize=False, positive=False,
precompute='auto', random_state=None, selection='cyclic', tol=0.0001,
verbose=False)
>>> # The estimator chose automatically its lambda:
>>> lasso.alpha_
0.01229...
这些估计值被称为与其对应的类似,其名称附加“CV”。
行使
在糖尿病数据集上,找到最优正则化参数α。
奖金:您可以信赖多少选择阿尔法?
from sklearn import datasets
from sklearn.linear_model import LassoCV
from sklearn.linear_model import Lasso
from sklearn.model_selection import KFold
from sklearn.model_selection import cross_val_score
diabetes = datasets.load_diabetes()
解决方案: 糖尿病数据集练习的交叉验证
无监督学习:寻求数据表示
聚类:将观察分组在一起
问题在聚类中解决了
鉴于虹膜数据集,如果我们知道有3种类型的虹膜,但没有访问分类学家来标注它们:我们可以尝试一个 聚类任务:将观察结果分成称为聚类的完全分离的组。
K-means聚类
请注意,存在很多不同的聚类标准和相关算法。最简单的聚类算法是 K-means。

>>> from sklearn import cluster, datasets
>>> iris = datasets.load_iris()
>>> X_iris = iris.data
>>> y_iris = iris.target
>>> k_means = cluster.KMeans(n_clusters=3)
>>> k_means.fit(X_iris)
KMeans(algorithm='auto', copy_x=True, init='k-means++', ...
>>> print(k_means.labels_[::10])
[1 1 1 1 1 0 0 0 0 0 2 2 2 2 2]
>>> print(y_iris[::10])
[0 0 0 0 0 1 1 1 1 1 2 2 2 2 2]
警告
绝对没有保证恢复地面真相。首先,选择正确的簇数很难。第二,该算法对初始化敏感,并且可以落入局部最小值,尽管scikit学习采用了几种技巧来缓解这个问题。
 |
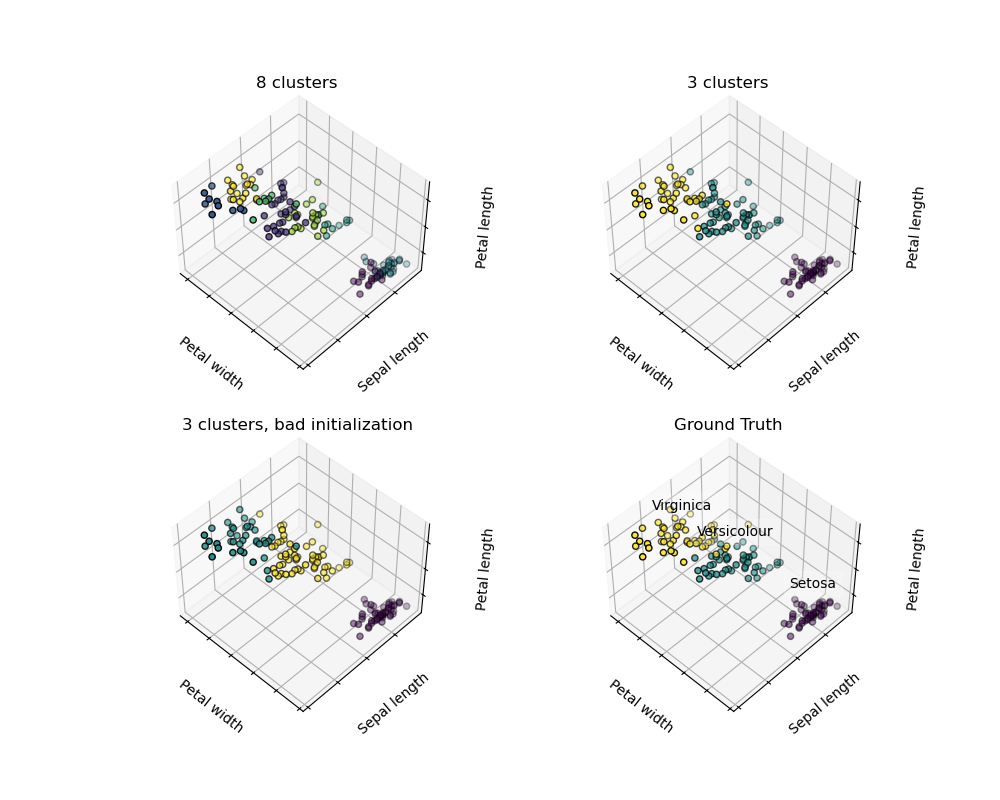 |
 |
| 初始化不好 | 8个群集 | 地面事实 |
不要过度解释聚类结果
应用实例:矢量量化
特别是集体聚焦和KMeans可以被看作是选择少量样本来压缩信息的一种方式。这个问题有时被称为 矢量量化。例如,这可以用于对图像进行后缀:
>>> import scipy as sp
>>> try:
... face = sp.face(gray=True)
... except AttributeError:
... from scipy import misc
... face = misc.face(gray=True)
>>> X = face.reshape((-1, 1)) # We need an (n_sample, n_feature) array
>>> k_means = cluster.KMeans(n_clusters=5, n_init=1)
>>> k_means.fit(X)
KMeans(algorithm='auto', copy_x=True, init='k-means++', ...
>>> values = k_means.cluster_centers_.squeeze()
>>> labels = k_means.labels_
>>> face_compressed = np.choose(labels, values)
>>> face_compressed.shape = face.shape
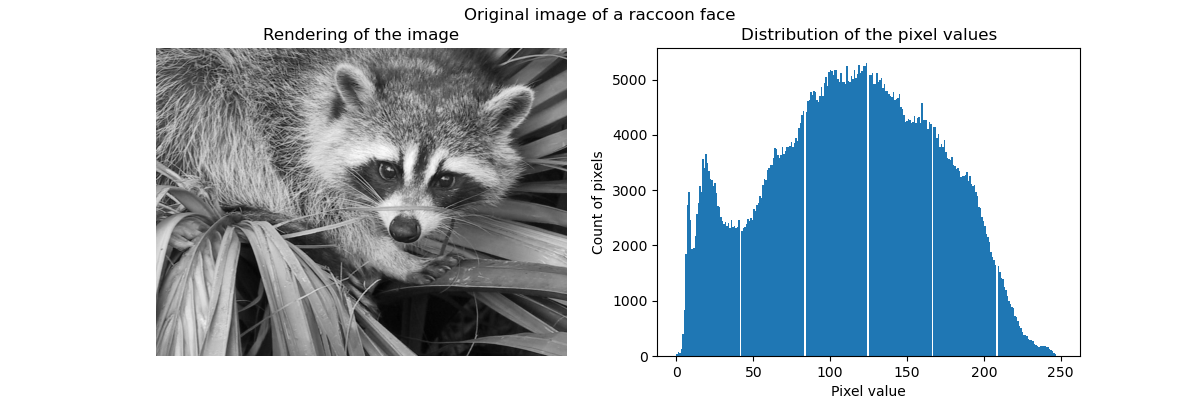 |
 |
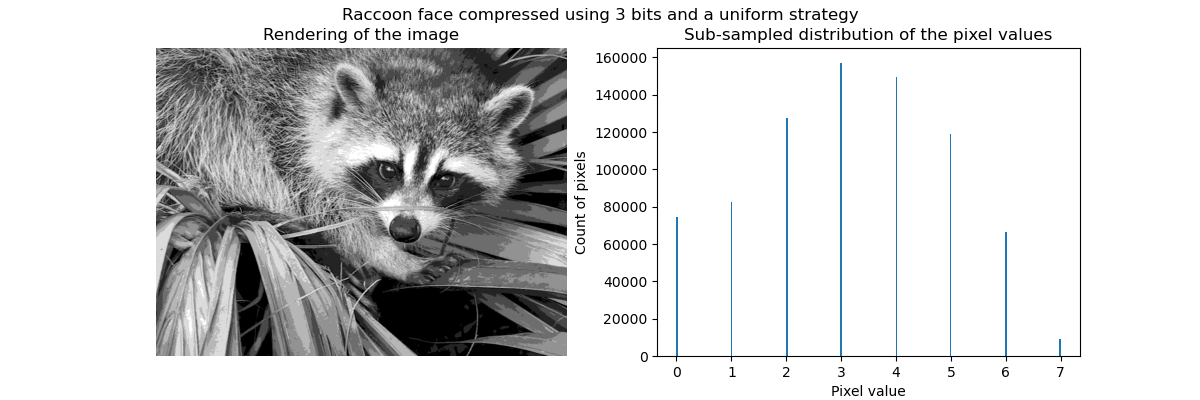 |
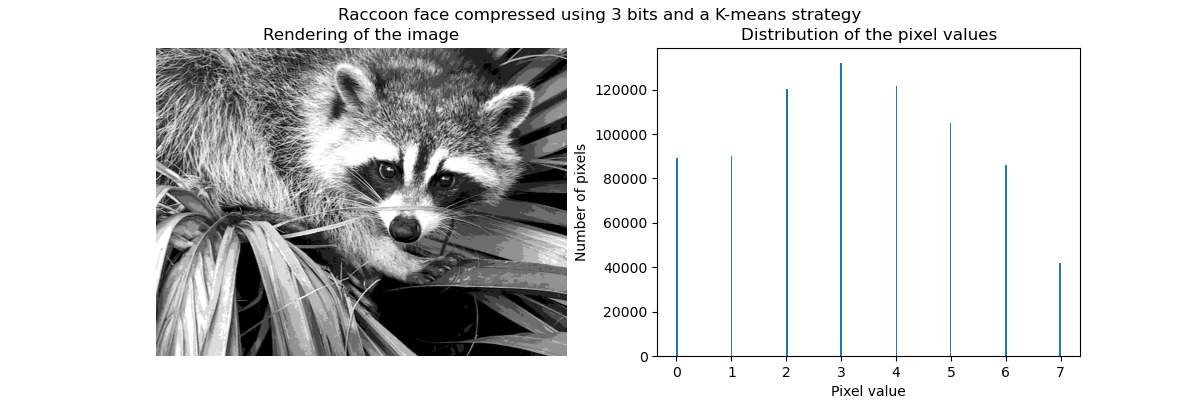 |
| 原始图像 | K均值量化 | 平仓 | 图像直方图 |
层次聚集:
甲分级聚类方法是一种类型的聚类分析的,其目的是建立聚类的层次结构。一般来说,这种技术的各种方法是:
- 集聚 - 自下而上的方法:每个观察在自己的集群中开始,集群被迭代地合并,以最小化连接标准。当感兴趣的集团仅由少数观察结果组成时,这种方法特别有意义。当簇的数量较大时,其计算效率要高于k-means。
- 分歧 - 自上而下的方法:所有的观察开始于一个群集,当层次结构向下移动时,迭代分裂。为了估计大量的群集,这种方法既缓慢(由于所有的观察开始于一个群集,它被递归地分解)和统计学上不合适。
连通约束聚类
通过聚集聚类,可以通过给出连通性图来指定哪些样本可以聚集在一起。scikit中的图表由它们的邻接矩阵表示。通常使用稀疏矩阵。这可以是有用的,例如,在聚类图像时检索连接的区域(有时也称为连接的组件):

import matplotlib.pyplot as plt
from sklearn.feature_extraction.image import grid_to_graph
from sklearn.cluster import AgglomerativeClustering
from sklearn.utils.testing import SkipTest
from sklearn.utils.fixes import sp_version
if sp_version < (0, 12):
raise SkipTest("Skipping because SciPy version earlier than 0.12.0 and "
"thus does not include the scipy.misc.face() image.")
###############################################################################
# Generate data
try:
face = sp.face(gray=True)
except AttributeError:
# Newer versions of scipy have face in misc
from scipy import misc
face = misc.face(gray=True)
# Resize it to 10% of the original size to speed up the processing
face = sp.misc.imresize(face, 0.10) / 255.
特征聚集
我们已经看到,稀疏性可以用来减轻维度的诅咒,即与特征数量相比,观察量不足。另一种方法是将类似特征合并在一起:特征聚集。这种方法可以通过在特征方向上聚类来实现,换句话说,聚类转置的数据。
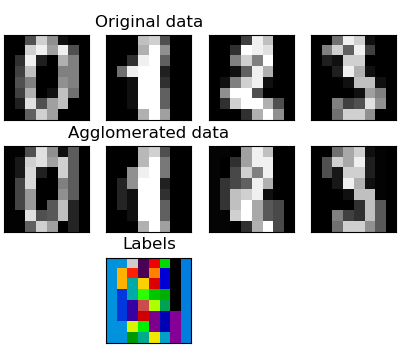
>>> digits = datasets.load_digits()
>>> images = digits.images
>>> X = np.reshape(images, (len(images), -1))
>>> connectivity = grid_to_graph(*images[0].shape)
>>> agglo = cluster.FeatureAgglomeration(connectivity=connectivity,
... n_clusters=32)
>>> agglo.fit(X)
FeatureAgglomeration(affinity='euclidean', compute_full_tree='auto',...
>>> X_reduced = agglo.transform(X)
>>> X_approx = agglo.inverse_transform(X_reduced)
>>> images_approx = np.reshape(X_approx, images.shape)
transform和inverse_transform方法
一些估计者会公开一种transform方法,例如降低数据集的维度。
分解:从信号到组件和加载
组件和装载
如果X是我们的多变量数据,那么我们要解决的问题是在不同的观察基础上重写:我们要学习加载L和一组组件C,使得X = LC。存在不同的标准来选择组件
主成分分析:
主分量分析(PCA)选择说明信号最大方差的连续分量。
上述观察点的点云在一个方向上是非常平坦的:三个单变量特征之一几乎可以使用另外两个方法精确计算。PCA查找数据不平坦的方向
用于变换数据时,PCA可以通过在主子空间上投影来降低数据的维数。
>>> # Create a signal with only 2 useful dimensions
>>> x1 = np.random.normal(size=100)
>>> x2 = np.random.normal(size=100)
>>> x3 = x1 + x2
>>> X = np.c_[x1, x2, x3]
>>> from sklearn import decomposition
>>> pca = decomposition.PCA()
>>> pca.fit(X)
PCA(copy=True, iterated_power='auto', n_components=None, random_state=None,
svd_solver='auto', tol=0.0, whiten=False)
>>> print(pca.explained_variance_)
[ 2.18565811e+00 1.19346747e+00 8.43026679e-32]
>>> # As we can see, only the 2 first components are useful
>>> pca.n_components = 2
>>> X_reduced = pca.fit_transform(X)
>>> X_reduced.shape
(100, 2)
独立成分分析:
独立组件分析(ICA)选择组件,使其负载的分布具有最大量的独立信息。它能够恢复 非高斯独立信号:
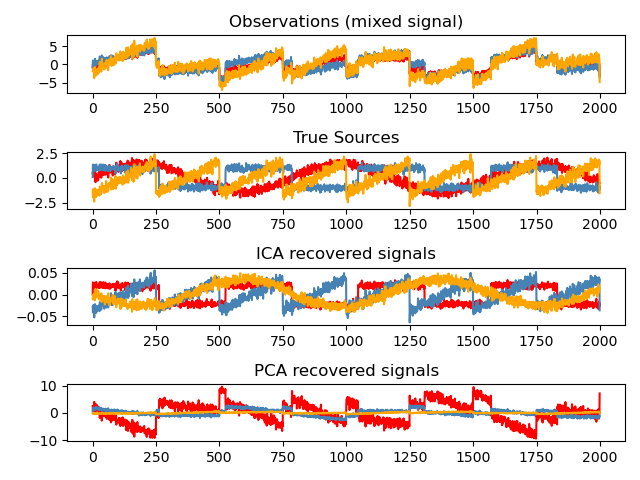
>>> # Generate sample data
>>> time = np.linspace(0, 10, 2000)
>>> s1 = np.sin(2 * time) # Signal 1 : sinusoidal signal
>>> s2 = np.sign(np.sin(3 * time)) # Signal 2 : square signal
>>> S = np.c_[s1, s2]
>>> S += 0.2 * np.random.normal(size=S.shape) # Add noise
>>> S /= S.std(axis=0) # Standardize data
>>> # Mix data
>>> A = np.array([[1, 1], [0.5, 2]]) # Mixing matrix
>>> X = np.dot(S, A.T) # Generate observations
>>> # Compute ICA
>>> ica = decomposition.FastICA()
>>> S_ = ica.fit_transform(X) # Get the estimated sources
>>> A_ = ica.mixing_.T
>>> np.allclose(X, np.dot(S_, A_) + ica.mean_)
True
把它放在一起
流水
我们已经看到一些估计器可以转换数据,一些估计器可以预测变量。我们还可以创建组合估计:
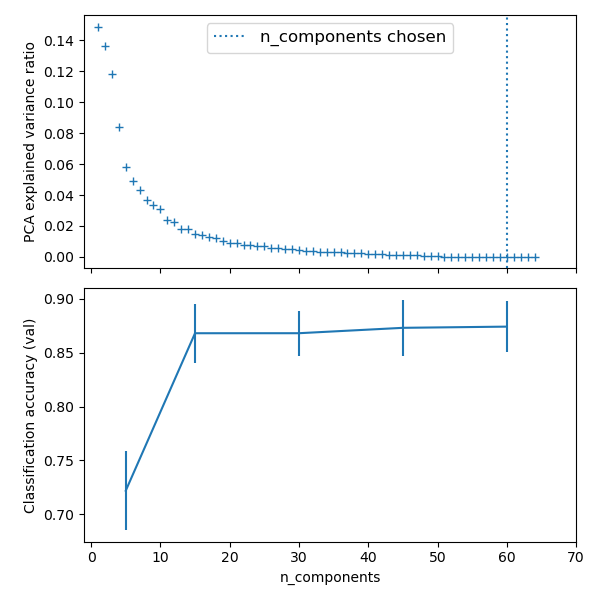
from sklearn import linear_model, decomposition, datasets
from sklearn.pipeline import Pipeline
from sklearn.model_selection import GridSearchCV
logistic = linear_model.LogisticRegression()
pca = decomposition.PCA()
pipe = Pipeline(steps=[('pca', pca), ('logistic', logistic)])
digits = datasets.load_digits()
X_digits = digits.data
y_digits = digits.target
###############################################################################
# Plot the PCA spectrum
pca.fit(X_digits)
plt.figure(1, figsize=(4, 3))
plt.clf()
plt.axes([.2, .2, .7, .7])
plt.plot(pca.explained_variance_, linewidth=2)
plt.axis('tight')
plt.xlabel('n_components')
plt.ylabel('explained_variance_')
###############################################################################
# Prediction
n_components = [20, 40, 64]
Cs = np.logspace(-4, 4, 3)
#Parameters of pipelines can be set using ‘__’ separated parameter names:
estimator = GridSearchCV(pipe,
dict(pca__n_components=n_components,
logistic__C=Cs))
estimator.fit(X_digits, y_digits)
plt.axvline(estimator.best_estimator_.named_steps['pca'].n_components,
linestyle=':', label='n_components chosen')
plt.legend(prop=dict(size=12))
使用特征面部识别
本例中使用的数据集是“野外标签面”的预处理摘录,也称为LFW:
http://vis-www.cs.umass.edu/lfw/lfw-funneled.tgz(233MB)
“”“
============================================= ====
使用特征面和SVM的面孔识别示例
=================================== ============
本示例中使用的数据集是
“野外标记的面孔” 的预处理摘录,也称为LFW_:
http://vis-www.cs.umass.edu/lfw/lfw-funneled.tgz(233MB)
.. _LFW:http://vis-www.cs.umass.edu/lfw/
数据集中最有代表人数的5个预期结果:
================== ============ ======= ========== === ====
精度回忆f1分数支持
================== ============ ======= === ======= =======
Ariel Sharon 0.67 0.92 0.77 13
Colin Powell 0.75 0.78 0.76 60
Donald Rumsfeld 0.78 0.67 0.72 27
George W Bush 0.86 0.86 0.86 146
Gerhard Schroeder 0.76 0.76 0.76 25
Hugo Chavez 0.67 0.67 0.67 15
托尼·布莱尔0.81 0.69 0.75 36
平均/合计0.80 0.80 0.80 322
================== ============ ======= ==== ==== =======
from __future__ import print_function
from time import time
import logging
import matplotlib.pyplot as plt
from sklearn.model_selection import train_test_split
from sklearn.model_selection import GridSearchCV
from sklearn.datasets import fetch_lfw_people
from sklearn.metrics import classification_report
from sklearn.metrics import confusion_matrix
from sklearn.decomposition import PCA
from sklearn.svm import SVC
print(__doc__)
# Display progress logs on stdout
logging.basicConfig(level=logging.INFO, format='%(asctime)s%(message)s')
###############################################################################
# Download the data, if not already on disk and load it as numpy arrays
lfw_people = fetch_lfw_people(min_faces_per_person=70, resize=0.4)
# introspect the images arrays to find the shapes (for plotting)
n_samples, h, w = lfw_people.images.shape
# for machine learning we use the 2 data directly (as relative pixel
# positions info is ignored by this model)
X = lfw_people.data
n_features = X.shape[1]
# the label to predict is the id of the person
y = lfw_people.target
target_names = lfw_people.target_names
n_classes = target_names.shape[0]
print("Total dataset size:")
print("n_samples: %d" % n_samples)
print("n_features: %d" % n_features)
print("n_classes: %d" % n_classes)
###############################################################################
# Split into a training set and a test set using a stratified k fold
# split into a training and testing set
X_train, X_test, y_train, y_test = train_test_split(
X, y, test_size=0.25, random_state=42)
###############################################################################
# Compute a PCA (eigenfaces) on the face dataset (treated as unlabeled
# dataset): unsupervised feature extraction / dimensionality reduction
n_components = 150
print("Extracting the top %d eigenfaces from %d faces"
% (n_components, X_train.shape[0]))
t0 = time()
pca = PCA(n_components=n_components, svd_solver='randomized',
whiten=True).fit(X_train)
print("done in %0.3fs" % (time() - t0))
eigenfaces = pca.components_.reshape((n_components, h, w))
print("Projecting the input data on the eigenfaces orthonormal basis")
t0 = time()
X_train_pca = pca.transform(X_train)
X_test_pca = pca.transform(X_test)
print("done in %0.3fs" % (time() - t0))
###############################################################################
# Train a SVM classification model
print("Fitting the classifier to the training set")
t0 = time()
param_grid = {'C': [1e3, 5e3, 1e4, 5e4, 1e5],
'gamma': [0.0001, 0.0005, 0.001, 0.005, 0.01, 0.1], }
clf = GridSearchCV(SVC(kernel='rbf', class_weight='balanced'), param_grid)
clf = clf.fit(X_train_pca, y_train)
print("done in %0.3fs" % (time() - t0))
print("Best estimator found by grid search:")
print(clf.best_estimator_)
###############################################################################
# Quantitative evaluation of the model quality on the test set
print("Predicting people's names on the test set")
t0 = time()
y_pred = clf.predict(X_test_pca)
print("done in %0.3fs" % (time() - t0))
print(classification_report(y_test, y_pred, target_names=target_names))
print(confusion_matrix(y_test, y_pred, labels=range(n_classes)))
###############################################################################
# Qualitative evaluation of the predictions using matplotlib
def plot_gallery(images, titles, h, w, n_row=3, n_col=4):
"""Helper function to plot a gallery of portraits"""
plt.figure(figsize=(1.8 * n_col, 2.4 * n_row))
plt.subplots_adjust(bottom=0, left=.01, right=.99, top=.90, hspace=.35)
for i in range(n_row * n_col):
plt.subplot(n_row, n_col, i + 1)
plt.imshow(images[i].reshape((h, w)), cmap=plt.cm.gray)
plt.title(titles[i], size=12)
plt.xticks(())
plt.yticks(())
# plot the result of the prediction on a portion of the test set
def title(y_pred, y_test, target_names, i):
pred_name = target_names[y_pred[i]].rsplit(' ', 1)[-1]
true_name = target_names[y_test[i]].rsplit(' ', 1)[-1]
return 'predicted: %s\ntrue: %s' % (pred_name, true_name)
prediction_titles = [title(y_pred, y_test, target_names, i)
for i in range(y_pred.shape[0])]
plot_gallery(X_test, prediction_titles, h, w)
# plot the gallery of the most significative eigenfaces
eigenface_titles = ["eigenface %d" % i for i in range(eigenfaces.shape[0])]
plot_gallery(eigenfaces, eigenface_titles, h, w)
plt.show()
 |
 |
| 预测 | 特征脸 |
数据集中最有代表人数的5个预期结果:
精准 回忆 f1 - 得分 支持
Gerhard_Schroeder 0.91 0.75 0.82 28
Donald_Rumsfeld 0.84 0.82 0.83 33
Tony_Blair 0.65 0.82 0.73 34
Colin_Powell 0.78 0.88 0.83 58
George_W_Bush 0.93 0.86 0.90 129
平均 / 总计 0.86 0.84 0.85 282
开放式问题:证券市场结构
我们可以预测Google在特定时间段内的股价变动吗?
模型选择:选择估计量及其参数
得分和交叉验证的分数
如我们所看到的,每个估计者都会公开一种score可以判断新数据的拟合质量(或预测)的方法。越大越好。
>>> 从 sklearn 导入 数据集, svm
>>> digits = 数据集。load_digits ()
>>> X_digits = 数字。数据
>>> y_digits = 数字。目标
>>> svc = svm 。SVC (C = 1 , kernel = 'linear' )
>>> svc 。fit (X_digits [:- 100 ], y_digits [:- 100 ])。得分(X_digits [ - 100 :], y_digits [ - 100 :])
0.97999999999999998
为了更好地测量预测精度(我们可以将其用作模型的拟合优势),我们可以连续地将数据分割成用于训练和测试的折叠:
>>> 将 numpy 作为 np
>>> X_folds = np 。array_split (X_digits , 3 )
>>> y_folds = np 。array_split (y_digits , 3 )
>>> scores = list ()
>>> for k in range (3 ):
... #我们使用'list'复制,以便稍后在“pop”
... X_train = 列表(X_folds )
.. 。X_test = X_train 。pop (k )
... X_train = np 。连接(X_train )
... y_train = list (y_folds )
... y_test = y_train 。pop (k )
... y_train = np 。连接(y_train )
... 得分。追加(SVC 。适合(X_train , y_train )。分数(X_test , y_test ))
>>> print (scores )
[0.93489148580968284,0.95659432387312182,0.93989983305509184]
这被称为KFold交叉验证。
交叉验证生成器
Scikit学习有一系列课程,可用于生成流行交叉验证策略的列车/测试指标列表。
它们公开了split一种接受要分割的输入数据集的方法,并为所选交叉验证策略的每次迭代产生列车/测试集合索引。
此示例显示了该split方法的示例用法。
>>> 从 sklearn.model_selection 导入 KFold , cross_val_score
>>> X = [ “a” , “a” , “b” , “c” , “c” , “c” ]
>>> k_fold = KFold (n_splits = 3 )
>>> for train_indices , test_indices in k_fold 。split (X ):
... print ('Train:%s | 测试:%s ' % (train_indices , test_indices ))
火车:[2 3 4 5] | 测试:[0 1]
火车:[0 1 4 5] | 测试:[2 3]
火车:[0 1 2 3] | 测试:[4 5]
然后可以轻松执行交叉验证:
>>> kfold = KFold (n_splits = 3 )
>>> [ svc 。fit (X_digits [ train ], y_digits [ train ])。得分(X_digits [ test ], y_digits [ test ])
... 用于 列车, 以k_fold 测试 。split (X_digits )] [0.93489148580968284,0.95659432387312182,0.93989983305509184]
交叉验证分数可以使用cross_val_score助手直接计算 。给定估计器,交叉验证对象和输入数据集,cross_val_score将数据重复分为训练和测试集,使用训练集训练估计器,并根据每次交叉验证迭代的测试集计算得分。
默认情况下,使用估计器的score方法来计算单个分数。
参考指标模块,了解更多可用评分方法。
>>> cross_val_score (svc , X_digits , y_digits , cv = k_fold , n_jobs = - 1 )
array([0.93489149,0.95659432,0.93989983])
n_jobs = -1表示将在计算机的所有CPU上分派计算。
或者,scoring可以提供参数以指定替代评分方法。
>>> cross_val_score (svc , X_digits , y_digits , cv = k_fold , ... scoring = 'precision_macro' ) array([0.93969761,0.95911415,0.94041254])交叉验证生成器
KFold (n_splits,shuffle,random_state) |
StratifiedKFold (n_iter,test_size,train_size,random_state) |
GroupKFold (n_splits,shuffle,random_state) |
| 将其分为K个折叠,K-1上的火车,然后在左侧测试。 | 与K-Fold相同,但保留每个折叠内的类分布。 | 确保同一组不在测试和训练集中。 |
ShuffleSplit (n_iter,test_size,train_size,random_state) |
StratifiedShuffleSplit |
GroupShuffleSplit |
| 根据随机排列产生列车/测试指标。 | 与shuffle split相同,但保留了每次迭代中的类分布。 | 确保同一组不在测试和训练集中。 |
LeaveOneGroupOut () |
LeavePGroupsOut (p)的 |
LeaveOneOut () |
| 使用组数组来对观察进行分组。 | 离开P组。 | 留下一个观察。 |
LeavePOut (p)的 |
PredefinedSplit |
| 留下P观察。 | 根据预定义的分割生成火车/测试指标。 |
行使

在数字数据集上,绘制SVC 具有线性内核的估计器的交叉验证分数作为参数的函数C(使用点对数网格,从1到10)。
导入 numpy的 是 NP
从 sklearn.model_selection 进口 cross_val_score
从 sklearn 进口 数据集, SVM
digits = 数据集。load_digits ()
X = 数字。数据
y = 数字。目标
svc = svm 。SVC (kernel = 'linear' )
C_s = np 。LOGSPACE (- 10 , 0 , 10 )
解决方案: 数字数据集练习的交叉验证
网格搜索和交叉验证的估计
网格搜索
scikit-learn提供了一个对象,给定的数据在参数网格中的估计器拟合期间计算分数,并选择参数以最大化交叉验证分数。该对象在构建期间使用估计器,并公开估计器API:
>>> 从 sklearn.model_selection 导入 GridSearchCV , cross_val_score
>>> Cs = np 。LOGSPACE (- 6 , - 1 , 10 )
>>> CLF = GridSearchCV (估计= SVC , param_grid = 字典(C ^ = 铯),
... n_jobs = - 1 )
>>> CLF 。fit (X_digits [:1000 ],
>>> #测试集上的预测性能不如火车上的设置
>>> clf 。得分(X_digits [ 1000 :], y_digits [ 1000 :])
0.943 ...
默认情况下,GridSearchCV使用三重交叉验证。然而,如果它检测到分类器被传递,而不是一个回归,它将使用分层3倍。
嵌套交叉验证
>>> cross_val_score (clf , X_digits , y_digits )
...
array([ 0.938 ...,0.963 ...,0.944 ...])
并行执行两个交叉验证循环:一个通过 GridSearchCV估计器进行设置gamma,另一个通过 cross_val_score测量估计器的预测性能。得出的分数是对新数据的预测分数的无偏估计。
警告
您不能使用并行计算来嵌套对象(n_jobs不同于1)。
交叉验证的估计
可以在逐个算法的基础上更有效地进行交叉验证来设置参数。这就是为什么,对于某些估计,scikit学习公开交叉验证:评估通过交叉验证自动设置其参数的估计器性能估计器:
>>> 从 sklearn import linear_model , datasets
>>> lasso = linear_model 。LassoCV ()
>>> 糖尿病 = 数据集。load_diabetes ()
>>> X_diabetes = 糖尿病。数据
>>> y_diabetes = 糖尿病。目标
>>> 套索。fit (X_diabetes , y_diabetes )
LassoCV(alphas = None,copy_X = True,cv = None,eps = 0.001,fit_intercept = True,
max_iter = 1000,n_alphas = 100,n_jobs = 1,normalize = False,positive = False,
precompute ='auto',random_state = None,selection ='cyclic',tol = 0.0001,
verbose = False)
>>> #自动选择其lambda:
>>> lasso 。alpha_
0.01229 ...
这些估计值被称为与其对应的类似,其名称附加“CV”。
行使
在糖尿病数据集上,找到最优正则化参数α。
奖金:您可以信赖多少选择阿尔法?
从 sklearn 进口 数据集
从 sklearn.linear_model 进口 LassoCV
从 sklearn.linear_model 进口 拉索
从 sklearn.model_selection 进口 KFold
从 sklearn.model_selection 进口 cross_val_score
糖尿病 = 数据集。load_diabetes ()
解决方案: 糖尿病数据集练习的交叉验证





 浙公网安备 33010602011771号
浙公网安备 33010602011771号