PCA是一种能够通过提取数据主成分达到数据降维目的的无监督算法。
因为数据之间(如自然图像的像素值)间都是存在冗余的,通过PCA可以将维度为256降到一个较低的近似向量。
通过一个2D降到1D的例子来理解一下PCA的原理。假设有如下一堆二维数据,我们通过SVD奇异值变换可以找到,代表这堆数据的两个方向(特征向量的方向,为什么是特征向量,特征值呢?)

怎么进行SVD变换呢?我们先计算这堆数据的协方差矩阵如下:
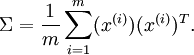 数据变化的主方向就是sigma的主特征向量,次方向就是sigma的次特征向量。接下来我们计算旋转后的数据(也就是说把数据投影到以这两个特征方向为坐标轴的坐标平面内)
数据变化的主方向就是sigma的主特征向量,次方向就是sigma的次特征向量。接下来我们计算旋转后的数据(也就是说把数据投影到以这两个特征方向为坐标轴的坐标平面内)
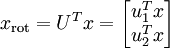
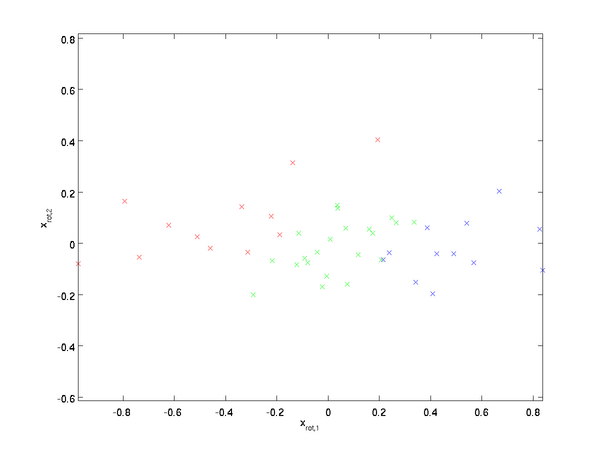
如图:
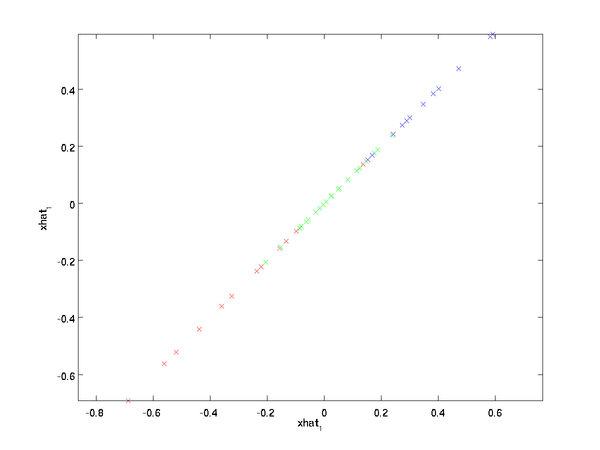
当我们只选取前面的k个主特征向量时,我们转换后就得到的是数据的主成分。PCA就是这样达到数据降维的。这里k=1,即选取主特征向量,得到数据的近似表示如下:、
一般我们根据近似误差的要求来选取k。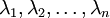 代表sigma协方差矩阵的特征值,一般地选择k保留百分之90的方差:
代表sigma协方差矩阵的特征值,一般地选择k保留百分之90的方差:
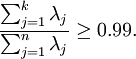
PCA经常和白化一起对图像进行预处理。PCA消除了输入特征间相关性。而白化之前要求的特征之间的低相关性要求正好就满足了。通过白化可以降低数据之间的冗余性。为了使得输入特征具有单位方差,我们利用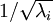 作为缩放因子对
作为缩放因子对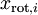 进行缩放。得到:
进行缩放。得到:
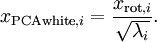 在实际中,我们往往给分母加入一个常量。
在实际中,我们往往给分母加入一个常量。
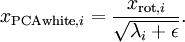
本文参照斯坦福UFLDL教程。http://deeplearning.stanford.edu/wiki/index.php/Exercise:PCA_in_2D
在matlab上实现这个例子代码如下:
close all %%=============================================================== %%step0:Load data % x is a 2*45 matrix,where the kth column x(:,k) corresponds to the % kth data point.Here we provide the code to load natural image into x. % You do not need to change the code below. x = load('pcaData.txt','-ascii'); figure(1); scatter(x(1,:),x(2,:)); title('Raw data'); %%===================================================================== %step1a:Implement PCA to obtain U u = zeros(size(x,1));%you need to campute this [n m] = size(x); x=x-repmat(mean(x,2),1,m);%预处理,均值为0 sigma = (1.0/m)*x*x' [u s v]=svd(sigma);%svd函数其实计算的是矩阵的奇异值和奇异矩阵,但就半正定矩阵来说,它们对应于特征值和特征向量, %那么矩阵U将包含sigma的特征向量(一个向量一列,从主向量开始排序),矩阵S对角线上的元素包含对应的特征值。矩阵V等于U的转制 hold on plot([0 u(1,1)],[0 u(2,1)])%画第一条线 plot([0 u(1,2)],[0 u(2,2)])%画第二条线 %==================================================================== %step1b:compute xRot,the projection on to the eigenbasis %Now,compute xRot by projecting the data on to the basis defined by %U.Visualize the points by performing a scatter plot. xRot = zeros(size(x)); xRot =u'*x; %Visualize the covariance matrix.You should see a line across the diagonal %against a blue backgroud. figure(2); scatter(xRot(1,:),xRot(2,:)); title('xRot'); %%============================================================================= %step2;Reduce the number of dimensions from 2 to 1. %Compute xRot again (this time project to 1 dimension) %then,compute xHat by projecting the xRot back onto the original axes to %see the effect of dimension reduction k=1;%Use k=1 and project the data onto the first eigenbasis xHat= zeros(size(x)); xHat = u*([u(:,1),zeros(n,1)]'*x); figure(3); scatter(xHat(1,:),xHat(2,:)); title('xHat'); %%=========================================================================== %step3:PCA Whitening %compute xPCAWhite and plot the results. epsilon = 1e-5; xPCAWhite = zeros(size(x)); xPCAWhite = diag(1./sqrt(diag(s)+epsilon))*u'*x; figure(4); scatter(xPCAWhite(1,:),xPCAWhite(2,:)); title('xPCAWhite'); %%=========================================================================== %step3:ZCA Whitening %Compute xZCAWhite and plot the results xZCAWhite = zeros(size(x)); xZCAWhite = u*diag(1./sqrt(diag(s)+epsilon))*u'*x; %------------------------------------- figure(5); scatter(xZCAWhite(1,:),xZCAWhite(2,:)); title('xZCAWhite');





 浙公网安备 33010602011771号
浙公网安备 33010602011771号