易基因:Nat Immunol/IF27.6:多组学分析揭示DNA低甲基化定义人组织调节性T细胞的表观遗传适应性程序及其血液再循环对应物
大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。
2025年7月16日,德国雷根斯堡大学Markus Feuerer教授和美因茨大学Michael Delacher 教授团队合作,通过整合人类皮肤组织和血液中调节性T细胞(Regulatory T cells,Treg细胞)的全基因组亚硫酸盐测序(WGBS)、染色质可及性测序(ATAC-seq)和转录组测序(RNA-seq)多组学数据,系统解析了人类皮肤组织和血液中Treg细胞的表观遗传特征。本研究发现皮肤Treg细胞表现出以DNA低甲基化为主的组织适应性呈现状态,尤其发生在转座子元件(TE)区域。进一步研究证实,血液中CCR8+ Treg细胞在DNA甲基化模式上与皮肤Treg高度相似,但在染色质可及性和基因表达上部分差异,提示其可能是从皮肤组织再循环至血液的Treg细胞。该研究首次利用多组学定义了人多组织Treg细胞的表观遗传适应性状态,并提出了CCR8+ Treg作为循环中组织来源Treg的甲基化标志。相关研究成果以“DNA hypomethylation traits define human regulatory T cells in cutaneous tissue and identify their blood recirculating counterparts”为题发表在免疫学相关领域顶刊《Nature immunology》。

英文标题:DNA hypomethylation traits define human regulatory T cells in cutaneous tissue and identify their blood recirculating counterparts
中文标题:DNA低甲基化表征定义了皮肤组织中的人类调节性T细胞,并鉴定出其血液再循环对应物
发表时间:2025年7月16日
发表期刊:Nature immunology
影响因子:IF27.6/Q1
技术平台:WGBS、ATAC-seq、RNA-seq(易基因金牌技术)
作者单位:雷根斯堡大学、美因茨大学等
DOI:10.1038/s41590-025-02210-x
组织中广泛存在的CD4+ Treg细胞发挥着至关重要的免疫调节和再生作用,但其表观遗传特征和分化机制仍未阐明。本研究对人的皮肤和血液Treg细胞进行全基因组DNA甲基化分析,并将其与染色质可及性和基因表达进行多组学关联分析,鉴定出调控皮肤Treg细胞的组织适应性程序。此外,研究发现转座元件亚家族是组织中Treg细胞低甲基化图谱的主要组分。有趣的是,基于T细胞抗原受体序列和DNA低甲基化同源性,本研究分析表明,血液CCR8+ Treg细胞中可能含有再循环的人类皮肤Treg细胞,而染色质可及性和基因表达差异分析表明,其组织适应程序在再循环过程中发生了一定程度的逆转。本研究结果为深入理解人类组织Treg细胞的生物学特性提供了见解,也为Treg细胞靶向治疗的安全性评估提供了重要的理论依据。
易基因相关拓展性产品案例
- 项目文章:山东大学基础医学院李雷教授团队微量WGBS揭示DNA甲基化调控斑马鱼造血干细胞发育的表观遗传机制
- CSC(IF20.5):剑桥大学团队利用微量WGBS等揭示DNMT3L在胎盘发育中的DNA甲基化调控机制
- 项目文章|微量WGBS+ACE-seq揭示卵巢早衰的人卵丘细胞DNA甲基化与羟甲基化表观基因组图谱
- 项目文章 | JGG:单细胞甲基化+转录组多组学分析揭示哺乳期母体低蛋白饮食对子代的跨代传递
- 项目文章:微量DNA甲基化分析揭示MeCP2在卵子发生和卵巢衰老中的表观遗传调控
- Cell|易基因微量DNA甲基化测序助力中国科学家成功构建胚胎干细胞嵌合体猴,登上《细胞》封面
研究方法
样本来源:
- 皮肤组织Treg细胞:来自7名健康女性捐赠者的皮肤组织。
- 血液Treg细胞:从10例健康女性血小板捐赠者的白细胞减除腔中分离外周血单核细胞(PBMCs),包括CCR8+ Treg、CD45RA+ naive Treg和常规T细胞(Tconv)。
- 脂肪组织Treg细胞:来自皮下脂肪,用于验证组织保守性。
表观多组学测序分析:
- WGBS(全基因组亚硫酸盐测序):用于全基因组DNA甲基化分析,覆盖约2.8M CpG位点,中位覆盖度2–7×。
- ATAC-seq(染色质可及性测序):利用公开数据及自行生成的数据,分析染色质开放区域。
- RNA-seq(转录组测序):分析基因表达差异,部分数据来源于公开数据库。
- 多组学整合:通过重叠DMRs、差异可及性峰值和差异表达基因,构建“DMR–peak–gene links”关联调控网络。
验证分析:
- T细胞受体(TCR)追踪:利用scRNA-seq/scTCR-seq数据,通过TCR克隆型重叠评估细胞来源同一性。
- 小鼠模型验证:使用光转换Kaede小鼠(Tg(CAG-Kaede)15Kgwa)追踪皮肤Treg细胞向引流淋巴结的迁移,并结合CCR8表达分析。
结果图形
(1)DNA低甲基化定义皮肤和血液CCR8+ Treg细胞
本研究首先通过WGBS技术对来自9例健康女性供者的血液naïve Treg细胞(CD45RA+CCR8-)、血液naïve Tconv细胞、血液CCR8+Treg细胞(CD45RA-CCR8+)、皮肤Treg细胞和皮肤Tconv细胞共5种CD4+ T细胞亚群的全基因组DNA甲基化进行深度测序分析。分析结果显示,皮肤Treg细胞和血液CCR8+ Treg细胞的全基因组DNA甲基化水平上表现出显著的DNA低甲基化表征,显著区别于血液CD45RA+ Treg/Tconv细胞,表明这两类细胞核心表观遗传程序共性。为精确定义细胞类型特异性甲基化特征,研究团队开发"signature region"算法用于筛选甲基化差异≥0.15、连续至少3个CpG、间隔<300 bp的区域。该算法共鉴定出182,204个特异性标记区域,低甲基化特征同时存在于皮肤Treg、皮肤Tconv和血液CCR8+ Treg细胞中,且这些区域主要富集于基因间区,远离CpG岛,表明低甲基化可能主要影响非编码调控元件。该结果首次从全基因组甲基化层面证实血液CCR8+ Treg细胞与组织Treg细胞的表观遗传共性。
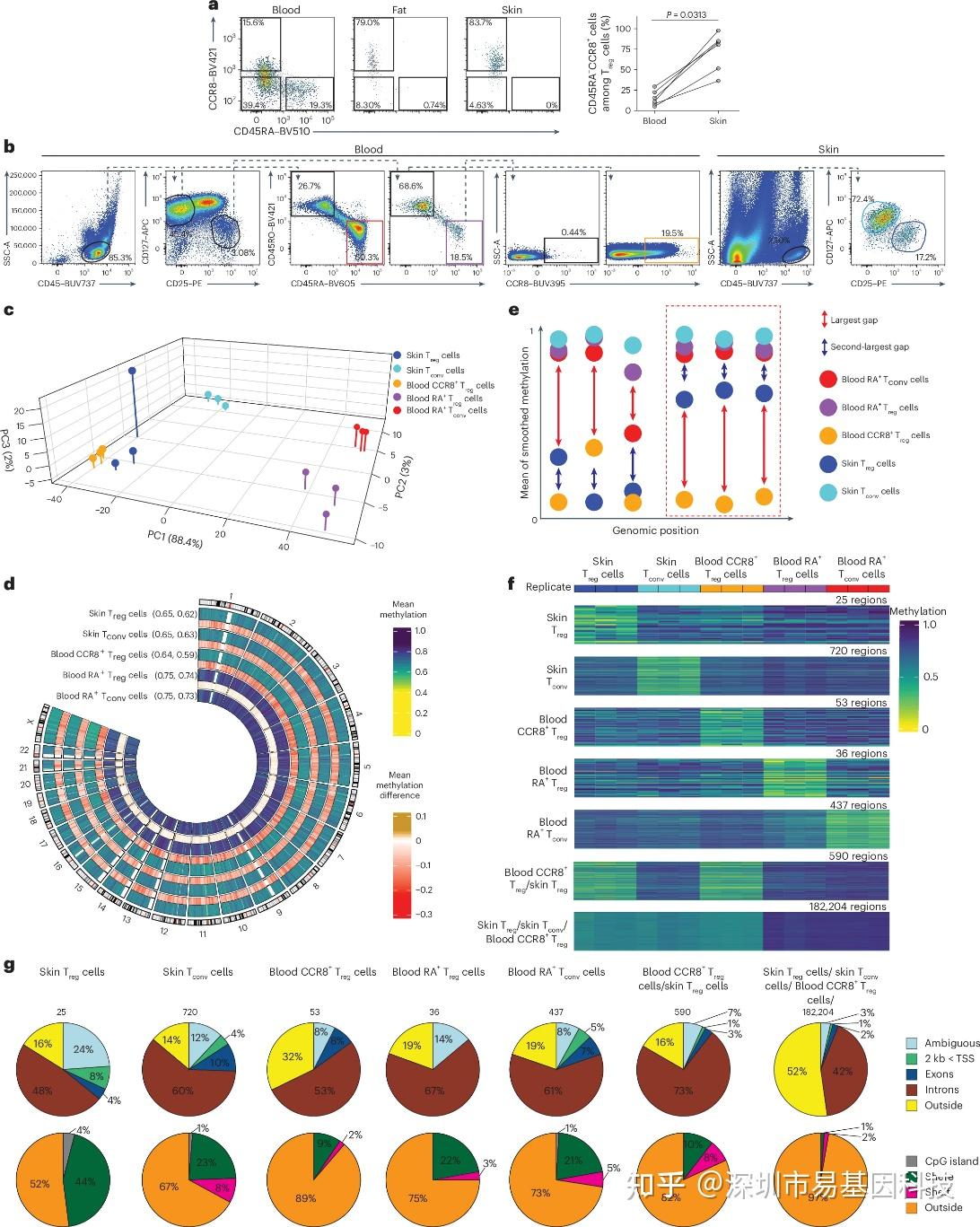
图1:皮肤Treg细胞和血液CCR8+ Treg细胞与血液CD45RA+ Treg细胞的DNA甲基化分析
a. 流式细胞术代表性图谱显示人类血液、脂肪和皮肤中CD4+CD127-CD25+ Treg细胞亚群的CD45RA-CCR8+ Treg细胞(左图),以及人类血液与皮肤中CD4+ CD127-CD25+ Treg细胞亚群的CD45RA-CCR8+ Treg细胞比例。
b. 分选策略示意图显示从1例健康女性供者中分离血液CD45RA+ Treg细胞、血液CD45RA+ Tconv细胞、血液CCR8+ Treg细胞、皮肤Tconv细胞和皮肤Treg细胞。
c. 对来自9例健康女性供者的血液和皮肤和血液中分离的血液CD45RA+ Treg细胞(RA+ Treg细胞)、血液CD45RA+ Tconv细胞(RA+ Tconv细胞)、血液CCR8+ Treg细胞、皮肤Tconv细胞和皮肤Treg细胞进行DNA甲基化主成分分析。
d. c中供者的人类血液CD45RA+ Treg细胞、血液CCR8+ Treg细胞、皮肤Tconv细胞和皮肤Treg细胞中按基因组位置显示的甲基化水平,以及与血液CD45RA+ Tconv细胞的差异分析。
e. 任意两种细胞类型之间最显著甲基化差异(红色箭头)提取细胞类型特征,从而筛选出特征区域。
f. e中所选细胞类型特征区域的DNA甲基化情况。
g. e中特征区域的细胞类型特征在由基因定义的基因组区间(上图)和CpG岛(下图)中的分布。
(2)多组学揭示差异甲基化区域-染色质开放峰-基因表达关联调控网络(DMR–peak–gene links)
为建立DNA甲基化与功能调控的直接联系,研究整合WGBS数据与已发表的scATAC-seq及bulk RNA-seq数据,构建了"DMR-peak-gene"关联分析调控网络,即一个基因组区域同时存在差异甲基化、差异可及性和关联基因差异表达。在比较血液CD45RA+ naïve Treg 和Tconv细胞,共鉴定出超3600个差异甲基化区域(DMRs),其中包含TNFRSF1B、TNFRSF9、IKZF2、IKZF4、FOXP3等经典Treg功能基因。进一步整合形成了151个DMR–peak–gene links,涉及73个基因。
在Treg表征位点(如FOXP3、TIGIT、IL2RA、CTLA4、TNFRSF1B)上,WGBS揭示的低甲基化模式与ATAC-seq检测的染色质开放及RNA-seq检测的高表达呈现显著负相关。此外,研究通过靶基因亚硫酸盐测序技术,在6例独立男性供者中验证了FOXP3、TIGIT、IL2RA、TNFRSF1B、LRRN3和SYTL3等位点的甲基化差异。这些关联分析揭示了甲基化变化如何通过染色质可及性调控基因表达,为理解Treg细胞功能调控提供了多组学证据链。
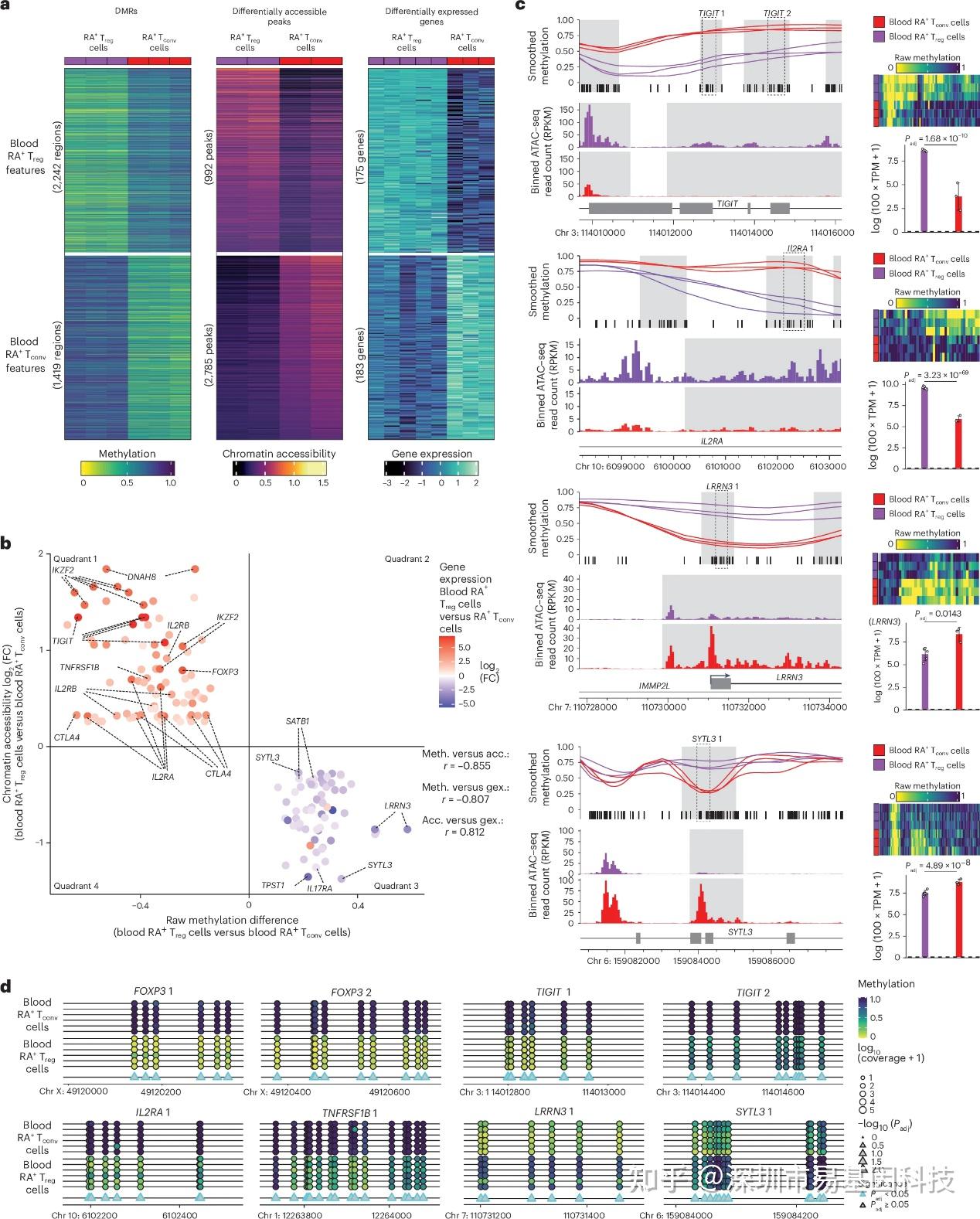
图2:多组学比较分析定义核心Treg细胞特征。
a. 血液CD45RA+ Treg细胞(RA+ Treg细胞)与血液CD45RA+ Tconv细胞(RA+ Tconv细胞)之间差异甲基化区域(左)、差异可及性peaks(中)和差异表达基因(右)的甲基化、染色质可及性和表达。
b. DMR–peak–gene关联分析(n=151)中差异甲基化、可及性和表达之间的相关性。
c. 血液CD45RA+ Treg细胞和血液CD45RA+ Tconv细胞中选定DMR–peak–gene关联的平滑甲基化(左上)、原始甲基化(右上)、染色质可及性(左下)和表达(右下)。
d. Taregt-BS测序显示选定区域血液CD45RA+ Treg细胞与血液CD45RA+ Tconv细胞的甲基化差异。
(3)皮肤Treg细胞组织适应具有多组学特征
为研究皮肤Treg细胞的组织特异性适应过程,研究团队鉴定出300,199个DMRs(其中298,457个为低甲基化)、8,914个差异可及性峰(3,192个高开放)和6,877个差异表达基因(4,049个高表达)。通过DMR-peak-gene关联分析,发现1,203个三联关联中,813个表现为低甲基化-高开放-高表达模式,涉及TNFRSF8(CD30)、RELB、CCR8、PRDM1和BATF等关键转录因子。WGBS数据显示,这些DMR甲基化水平变化程度远高于染色质可及性变化,提示DNA甲基化可能是促进组织适应的"主开关"。功能富集分析表明,低甲基化关联基因显著富集于IL-2-STAT5信号、TNF信号、TGFβ信号等Treg细胞核心通路。值得注意的是,将皮肤Treg特征与脂肪组织Treg数据交叉验证,发现脂肪Treg细胞中共有绝大多数低甲基化特征,证明了WGBS鉴定的表观遗传适应程序在不同组织的Treg细胞间具有保守性。该结果揭示,组织Treg细胞的适应过程本质上是一个以DNA去甲基化为主导的大规模表观遗传重编程事件,而非简单的转录因子诱导表达。
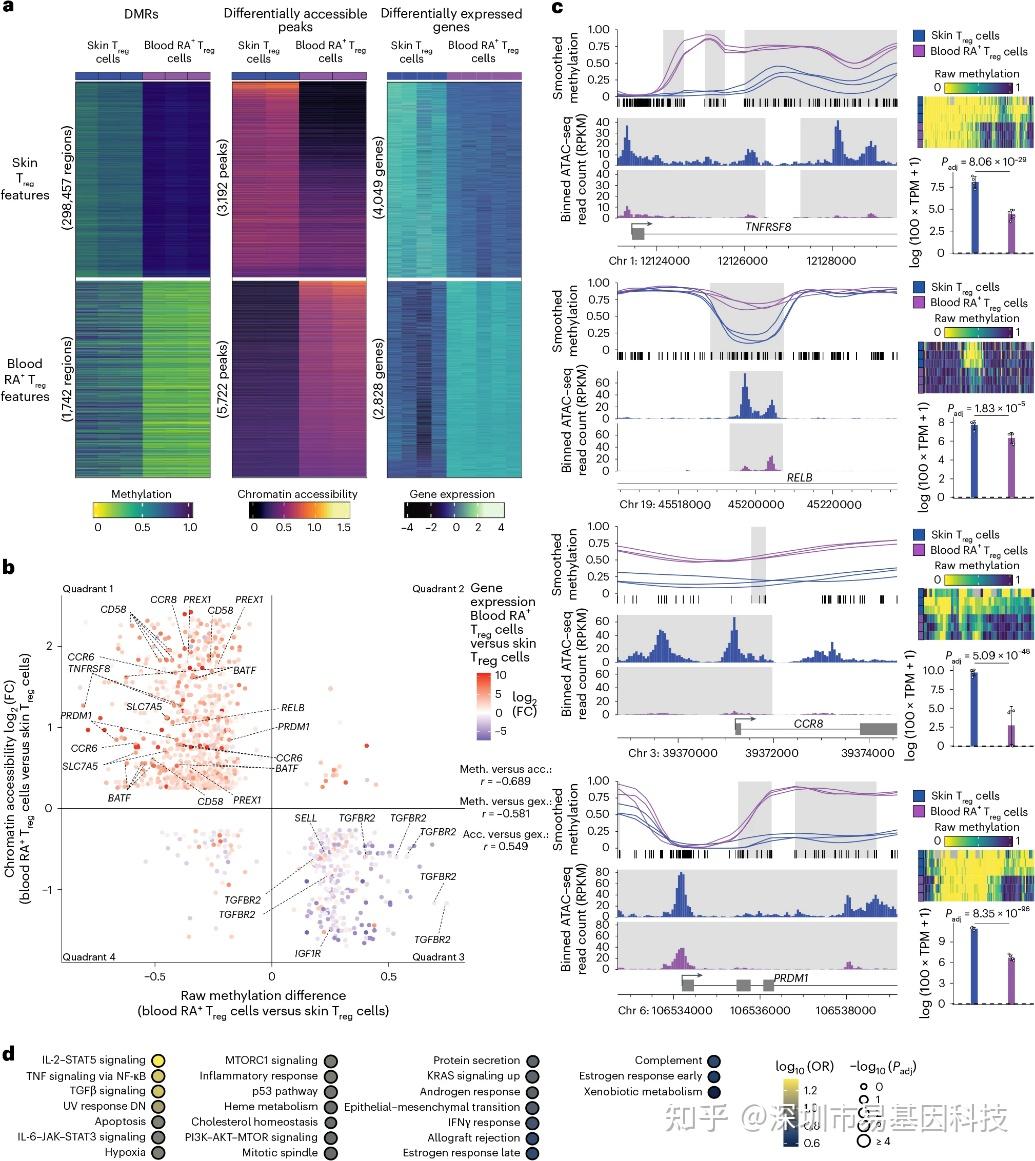
图3:DMR–peak–gene关联分析定义皮肤Treg细胞发育的多组学表征。
a. 皮肤Treg细胞与血液CD45RA+ Treg细胞(血液RA+ Treg细胞)之间差异甲基化区域(左)、差异可及性峰(中)和差异表达基因(右)的甲基化、染色质可及性和表达情况。
b. DMR–peak–gene关联(n=1,203个)中差异甲基化、可及性和基因表达之间的相关性。
c. 所选DMR–peak–gene关联分析显示在皮肤Treg细胞和血液CD45RA+ Treg细胞中的平滑甲基化(左上)、原始甲基化(右上)、染色质可及性(左下)和基因表达(右下)。
d. 多组学分析显示皮肤Treg细胞特征中基因的标记基因集富集情况。
(4)bZIP和bHLH motif的甲基化模式标记皮肤Treg细胞
通过解析DMRs中的转录因子结合位点,研究发现在皮肤Treg细胞低甲基化DMRs和高可及性峰中,bZIP转录因子(如BATF、Jun-AP1)的结合motifs中显著富集。而bHLH转录因子(如c-Myc、n-Myc、USF1)motif仅富集于低甲基化DMRs,未在可及性峰值中富集。这些结果表明,bZIP因子可能在皮肤Treg细胞适应中同时受甲基化与可及性调控,而bHLH因子可能受甲基化调控进而发挥功能。
为验证这些低甲基化位点功能,研究选取MLPH和GNA11位点的c-Myc结合位点,运用CRISPR激活系统(CRISPRa)进行靶向激活,结果显示这两个基因表达显著上调,验证了c-Myc结合位点低甲基化与邻近基因(如MLPH、GNAI1)表达上调的相关性。这一功能实验直接证明WGBS鉴定的低甲基化区域具有增强子活性,且其调控作用依赖于精确的甲基化状态。
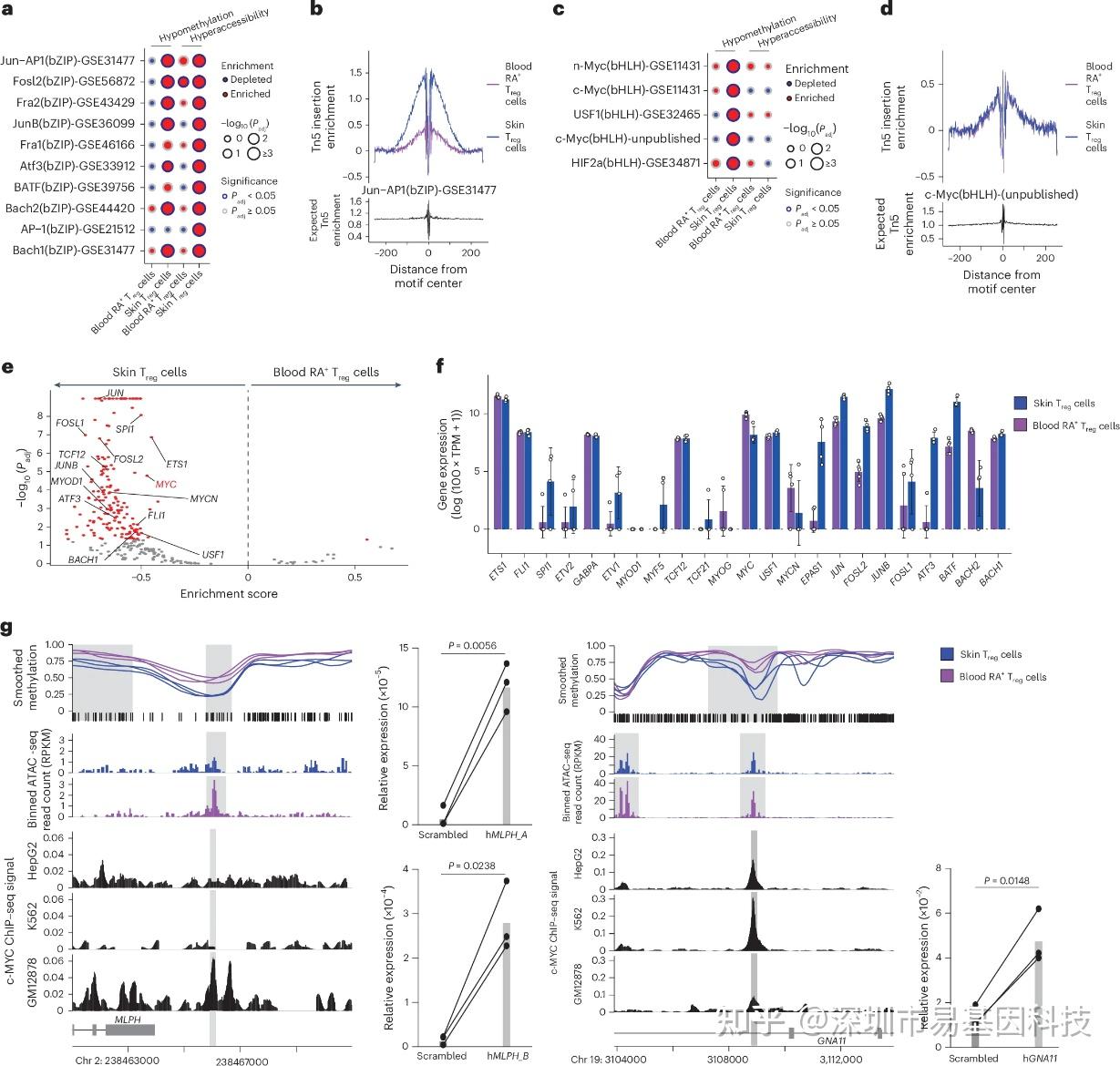
图4:皮肤Treg细胞发育过程中bZIP和bHLH转录因子活性与其结合位点的低甲基化相关
(5)血液CCR8+ Treg细胞与皮肤Treg细胞的DNA甲基化模式相似
研究将血液CCR8+ Treg细胞与皮肤Treg与血液naïve Treg的甲基化谱进行重叠分析,发现在97%的DMRs中,CCR8+ Treg细胞的甲基化水平更接近皮肤Treg细胞。然而,在染色质可及性(约60%)和基因表达(约45%)谱中,相似性显著降低。这种"甲基化-可及性-表达"的梯度递减现象提示,再循环Treg细胞在离开组织后,血液CCR8+ Treg细胞可能保留了组织Treg的甲基化“记忆”,但在循环过程中的部分可及性与表达程序被逆转。
对BATF、PREX1和SPRED2等位点的进一步分析显示,尽管这些区域的甲基化状态在CCR8+ Treg中维持低水平,但染色质开放状态未维持,已趋近初始Treg,揭示甲基化作为更稳定表观遗传标记的特性。小鼠Kaede光转化实验进一步证实,65%的迁出皮肤引流淋巴结的Treg细胞表达CCR8,而原位Treg仅30%表达,从体内动态角度验证了CCR8+ Treg细胞的组织来源。该结果为确定血液CCR8+ Treg细胞作为组织驻留Treg细胞的循环对应物提供了证据。
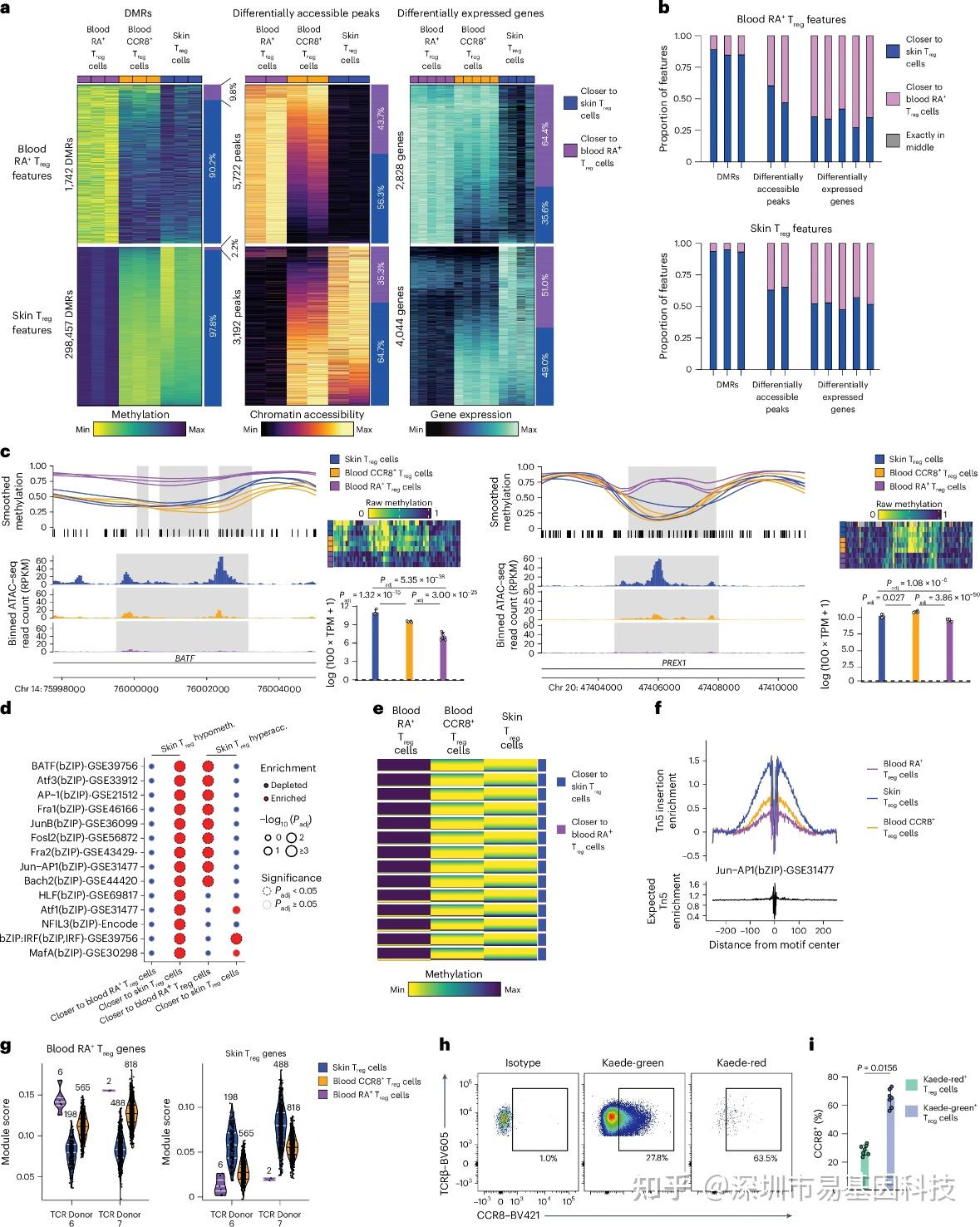
图5:皮肤Treg细胞与血液CCR8+ Treg细胞的DNA甲基化模式具有相似性
(6)转座元件(TF)低甲基化是组织Treg细胞的标记
转座子元件(TE)在人类基因组中占比约40–50%,常被DNA甲基化沉默。本研究发现,在皮肤Treg细胞中,SINE、LTR、DNA等TE亚家族(如LTR18B、HUERS-P2-int、MIR、L2a)在低甲基化DMRs中显著富集。直接分析TE插入位点水平显示,皮肤Treg细胞中LTR、SINE、DNA类TE的甲基化水平显著低于血液CD45RA+ Treg。这些TE插入位点常位于TNFRS8、PREX1、PRDM1、BATF等皮肤Treg特征基因内部或邻近区域,提示TE低甲基化可能通过调控邻近基因表达参与组织适应性。
将血液CCR8+ Treg细胞纳入比较后,发现这些细胞同样富集TE低甲基化特征,且程度与皮肤Treg类似,但未在可及性中富集,进一步支持TE低甲基化是组织Treg细胞的稳定表观遗传印记。
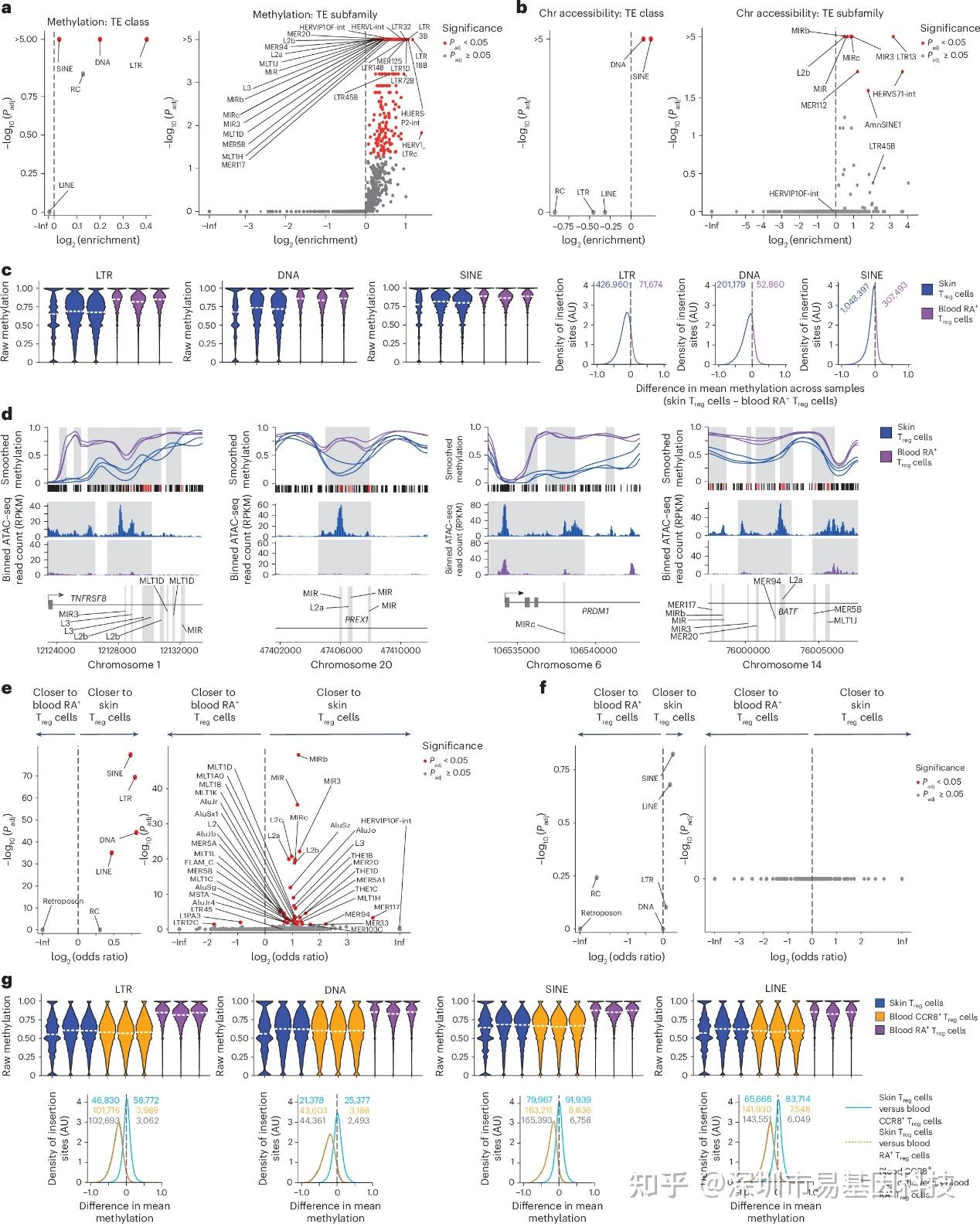
图6:转座元件在皮肤Treg细胞和血液CCR8+ Treg细胞中发生低甲基化
(7)TE亚家族在皮肤Treg细胞中获得RNA表达
为验证TE低甲基化是否导致其转录激活,研究进一步发现,部分TE亚家族在皮肤Treg细胞中转录上调,HERVIP10F-int和LTR45B在皮肤Treg细胞中显著高表达。甲基化数据显示,这两个TE亚家族的插入位点在皮肤和脂肪Treg细胞中均呈现显著低甲基化,且在血液CCR8+ Treg细胞中同样保守。表1列出了5个最显著低甲基化的HERVIP10F-int和LTR45B插入位点,其中,染色体4上的一个HERVIP10F-int插入位点在皮肤Treg中甲基化降低达74%。这表明TE不仅是低甲基化的被动载体,还可能通过转录活性参与Treg细胞的组织适应调控。该结果不仅为TE参与免疫调控提供了人类原代细胞证据,也暗示TE再激活可能是组织Treg细胞获得组织修复功能的分子基础之一。
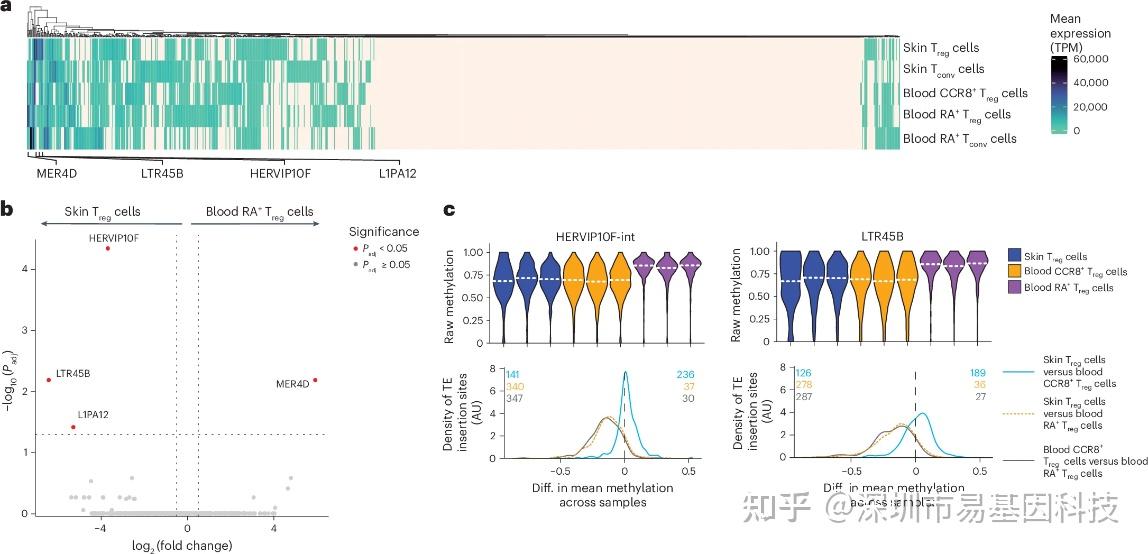
图7:多个TE亚家族在皮肤Treg细胞中转录上调
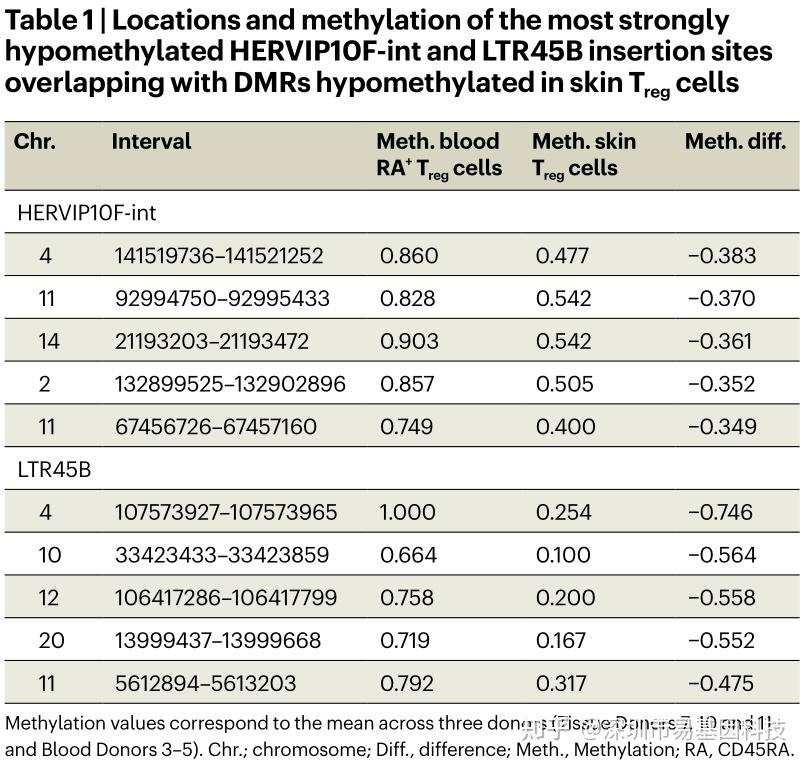
表1:与皮肤Treg细胞低甲基化DMR重叠的低甲基化最显著HERVIP10F-int和LTR45B插入位点位置及甲基化水平
结论和启示
本研究建立了人类多组织Treg细胞高分辨率DNA甲基化图谱,揭示以全基因组低甲基化和TE再激活为核心的组织适应性程序,并多层次证实血液CCR8+ Treg细胞是组织驻留Treg的再循环对应物。主要结论包括:
(1)DNA甲基化是定义组织Treg细胞身份稳定表观遗传标记;
(2)TE低甲基化是组织Treg特征的重要组分;
(3)血液CCR8+ Treg细胞保留组织甲基化记忆,但转录和染色质状态部分恢复初始样特征;
(4)靶向CCR8+ Treg细胞的治疗需谨慎评估对组织稳态的影响。
WGBS作为本研究中全基因组DNA甲基化图谱的核心关键技术:1.鉴定出组织与血液Treg细胞的甲基化差异;2.发现TE区域的广泛低甲基化;3.通过甲基化相似性推断细胞起源与迁移关系;4.整合多组学数据建立DMR–peak–gene调控网络。该技术在此类研究中的优势在于其全面性与分辨率,未来可广泛适用于追溯细胞发育与迁移轨迹、解析复杂组织中细胞亚型的表观遗传异质性和发现非编码区域(如TE)在细胞功能中的调控作用等。
目前,易基因科技有限公司在DNA甲基化修饰等表观遗传检测领域拥有一系列国际先进技术,可对高通量单细胞甲基化组学研究,微量DNA甲基化测序,ctDNA、FFPE等严重降解痕量DNA甲基化检测等,提供多种有效解决方案。低成本、高通量的单细胞甲基化组学测序技术建立,将为研究者对不同时间和空间的单细胞表观修饰研究奠定基础。
1)微量细胞或单细胞全基因组甲基化测序(Micro DNA-WGBS)DNA起始量:
单细胞/100-1000个细胞
1ng基因组DNA
90%以上基因组CG覆盖
2)微量细胞或DNA简化基因组甲基化测序(Micro DNA-RRBS)DNA起始量:
1ng基因组DNA;
10-20M有效CG位点覆盖;
10-20G测序数据量;
3)微量cfDNA简化基因组甲基化测序(cfDNA-RBS)
100ul血浆或1ng cfDNA
10M有效CG位点覆盖
15-20G测序数据量
易基因DNA甲基化整体研究方案
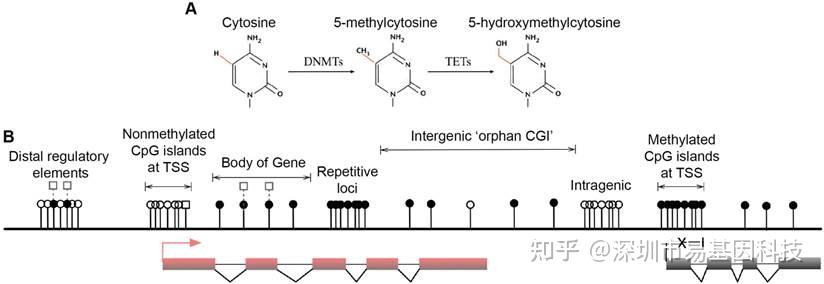
DNA甲基化是一种表观遗传修饰,指DNA分子在DNA甲基转移酶的作用下将甲基选择性地添加到特定碱基上的过程。主要发生在胞嘧啶的CpG位点,也可以在非CpG位点和CpG岛中发生。DNA甲基化参与调控基因表达、X染色体失活、基因印记、胚胎发育、细胞分化、肿瘤发生以及维持基因组稳定性。
参考文献:
Beumer N,et al. DNA hypomethylation traits define human regulatory T cells in cutaneous tissue and identify their blood recirculating counterparts. Nat Immunol. 2025 Jul 16. doi: 10.1038/s41590-025-02210-x.
相关阅读:
1.CSC(IF20.5):剑桥大学团队利用微量WGBS等揭示DNMT3L在胎盘发育中的DNA甲基化调控机制
2.项目文章|微量WGBS+ACE-seq揭示卵巢早衰的人卵丘细胞DNA甲基化与羟甲基化表观基因组图谱
3.项目文章 | JGG:单细胞甲基化+转录组多组学分析揭示哺乳期母体低蛋白饮食对子代的跨代传递
4.项目文章:微量DNA甲基化分析揭示MeCP2在卵子发生和卵巢衰老中的表观遗传调控
5.Cell|易基因微量DNA甲基化测序助力中国科学家成功构建胚胎干细胞嵌合体猴,登上《细胞》封面
6.项目文章|微量转录组测序揭示TET酶在动物卵母细胞成熟和孤雌胚胎发育中的作用机制
7.项目文章:山东大学基础医学院李雷教授团队微量WGBS揭示DNA甲基化调控斑马鱼造血干细胞发育的表观遗传机制




 浙公网安备 33010602011771号
浙公网安备 33010602011771号