第八章——降维(Dimensionality Reduction)
机器学习问题可能包含成百上千的特征。特征数量过多,不仅使得训练很耗时,而且难以找到解决方案。这一问题被称为维数灾难(curse of dimensionality)。为简化问题,加速训练,就需要降维了。
降维会丢失一些信息(比如将图片压缩成jpeg格式会降低质量),所以尽管会提速,但可能使模型稍微变差。因此首先要使用原始数据进行训练。如果速度实在太慢,再考虑降维。
8.1 维数灾难(The Curse of Dimensionality)
我们生活在三维空间,连四维空间都无法直观理解,更别说更高维空间了(wiki有关四维空间的介绍,以及油管上的一个视频,将四维空间展开为三维空间)。高维空间和低维空间相比,还是用很大区别的。比如一个单位正方形,只有大概0.4%的部分是距离边界0.001以内的(这部分边缘的面积大概是$0.001 \times 1 \times 4 = 0.004$,占总体面积的0.4%)。但是在一个一万维的单位超立方体中,这一概率却变成了99.999999%,绝大多数点都在距离某一维度很近的地方。一个有趣的事实是,人类有许多不同的属性,你认识的所有人都可能是某一特征的极端分子(比如咖啡里的放糖量)。
还有一个更麻烦的区别:如果在单位正方形中任意选取两点,其距离平均大概是0.52(这道概率统计的习题,我已经不记得怎么求解了)。在单位立方体中这一距离是0.66。而在1000000维单位超立方体中,这一距离就增大到了408.25(大概$\sqrt{1000000/6}$)。这说明高维数据集很可能是相当稀疏的,样本实例间距离很大,预测是新的样本距离训练集样本的距离也很大,预测可信度远低于地位数据集。简单来说,高维数据集很容易过拟合。
理论上,维数灾难的一个解决方案是增加样本数量,从而使训练集达到足够的密度。可是这在实践中并不可行,因为其复杂度是指数级的。
8.2 主要降维方法(Main Approaches for Dimensionality Reduction)
8.2.1 投影(Projection)
如图8-2所示,可将三维数据集投影到合适的二维平面,
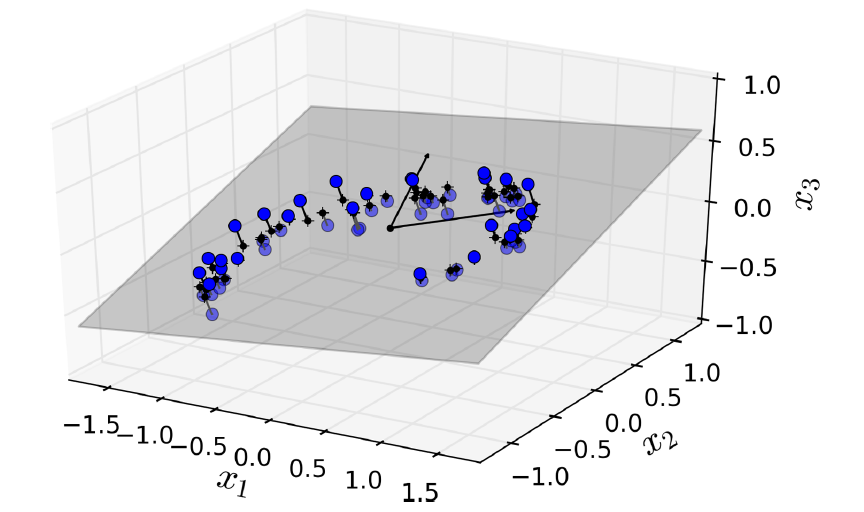
图8-2. 几乎位于同一平面的3D数据集
投影后的新数据集如图8-3所示。
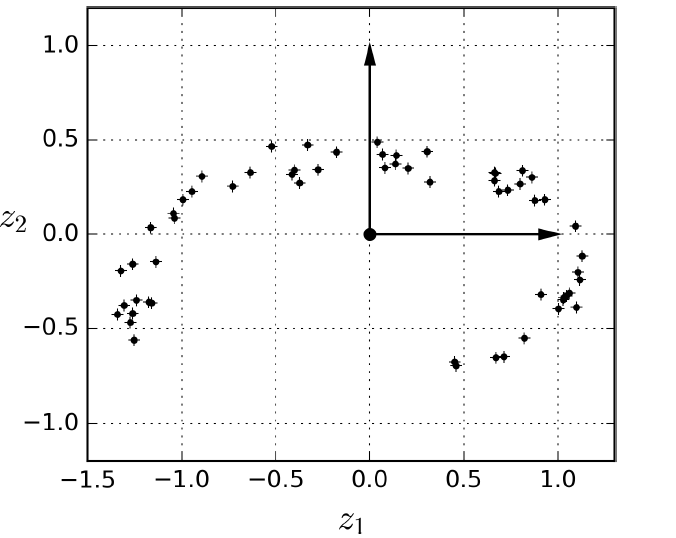
图8-3. 投影和的2D数据集
但有时候,投影是不好使的,例如图8-4所示的蛋糕卷数据集,尽管该数据集也是近似位于一个二维平面的(该数据集正确的降维方式是将蛋糕卷展开的二维平面)。
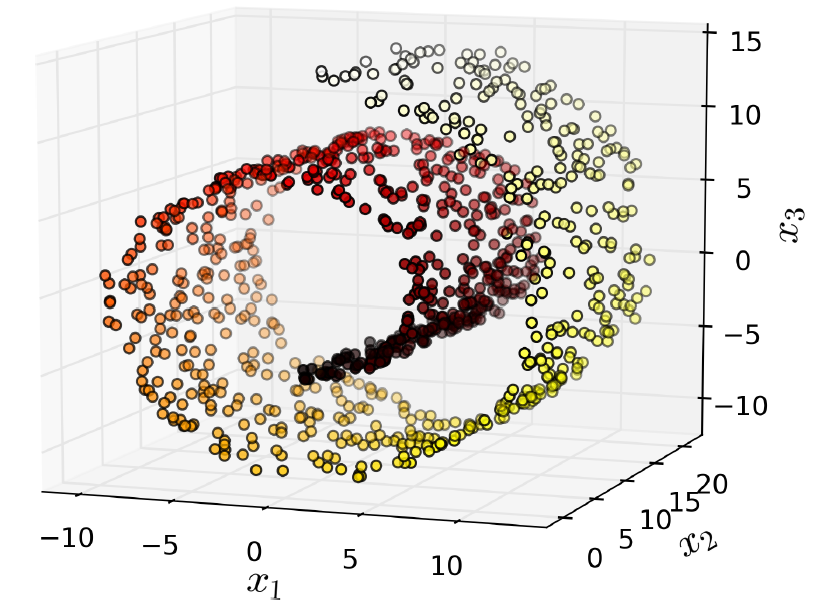
8-4. Swiss roll数据集
8.2.2 流形学习(Manifold Learning)
上面的蛋糕卷数据集就是一个2D流形。简单来讲,2D流形是一个在更高维空间中扭曲的2D形状。 当然,2D流形可以推广到n维流形。
许多降维算法就是对数据集所处于的流形建模,这被称作流行学习。
8.3 主成分分析(PCA)
主成分分析(Principal Component Analysis)是目前最受欢迎的降维算法。该算法首先找到距离数据集最近的低维超平面,然后就数据集投射到该超平面。
8.3.1 保留方差(Preserving the Variance)
将数据集投影到低维超平面之前,首先要选择合适的超平面。如图8-7所示,左侧是一个二维数据集,以及三个超平面(一维超平面)。右侧是数据集在相应超平面做投影后得到的新数据集。
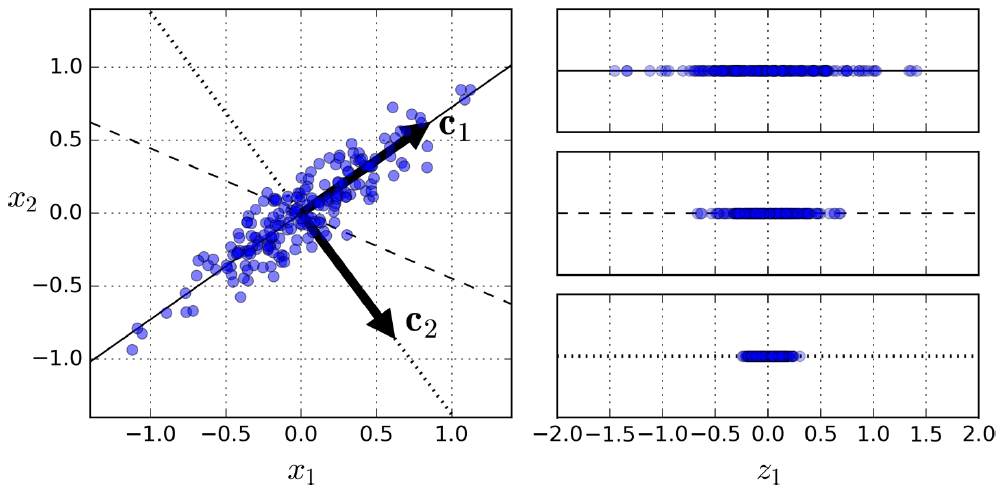
图8-7. 选择合适的子空间
很明显应该选择那条实心直线作为超平面,因为投影后新的数据集方差最大,损失的信息最少。此外,该超平面对应的新数据集,与原始数据集的欧氏距离最小。
8.3.2 主成分(Principal Components)
PCA可以识别出最大程度地保留训练数据集差异(variance)的坐标轴,也就是图8-7中的实线。然后找出第二个坐标轴,最大程度地保留剩下的差异,也就是图中与实线正交的虚线。用单位向量表示第$i$个坐标轴,这被称作第$i$个主成分。
可以使用奇异值分解(Singular Value Decomposition,SVD)得到主成分,$X = U \cdot \Sigma \cdot V^T$。
主成分矩阵:
\begin{align*}
V^T = \begin{pmatrix}
| &| & &| \\
c_1 &c_2 &\cdots &c_n \\
| &| & &|
\end{pmatrix}
\end{align*}
NumPy的svd() 函数可以进行奇异值分解:
X_centered = X - X.mean(axis=0) U, s, V = np.linalg.svd(X_centered)
PCA假设数据集是以原点为中心的,只不过Scikit-Learn的PAC类会自动进行中心化。
8.3.3 投影到d维(Projecting Down to d Dimensions)
最佳超平面由前d个主成分向量组成,将训练集投影到该超平面即可,代码如下:
W2 = V.T[:, :2] X2D = X_centered.dot(W2)
8.3.4 使用Scikit-Learn
from sklearn.decomposition import PCA pca = PCA(n_components = 2) X2D = pca.fit_transform(X)
8.3.5 可解释方差比例(Explained Variance Ratio)
>>> print(pca.explained_variance_ratio_) array([ 0.84248607, 0.14631839])
这说明第一个主成分承担了84.2%的差异,第二个主成分承担了14.6%的差异。
8.3.6 维数选择
与其武断地选择多杀维,不如选择足够保留大部分差异(比如95%)的维数。以下代码获取足以保留95%差异的代码:
pca = PCA() pca.fit(X) cumsum = np.cumsum(pca.explained_variance_ratio_) d = np.argmax(cumsum >= 0.95) + 1
然后设置n_components=d重新执行PCA:
pca = PCA(n_components=0.95) X_reduced = pca.fit_transform(X)
另一个选择是以维数为自变量,可释方差(explained variance)为因变量,画出函数图形:
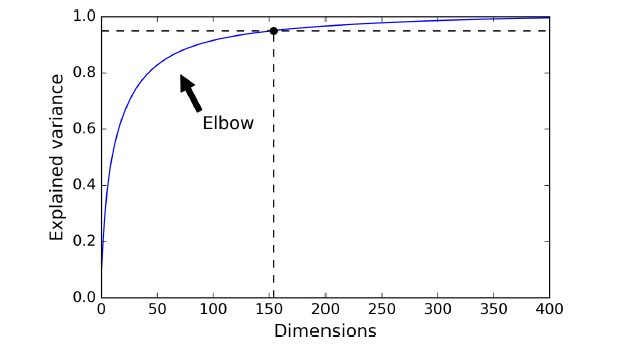
8.3.7 PCA用于压缩
在MNIST数据集应用PCA,保留95%的差异,仅需150个特征,远小于原始的784个特征,这是一个不错的压缩率。压缩之后还可以解压缩到784维,但这并不能得到原始数据,只是跟选择数据很接地,毕竟压缩做投影的时候丢失了一些信息。
8.3.8 Incremental PCA
感觉类似于最小批梯度下降
8.3.9 随机PCA(Randomized PCA)
可以加速运算
8.4 核PCA(Kernel PCA)
核PCA就是将核技巧应用于PCA,代码如下:
from sklearn.decomposition import KernelPCA rbf_pca = KernelPCA(n_components = 2, kernel="rbf", gamma=0.04) X_reduced = rbf_pca.fit_transform(X)
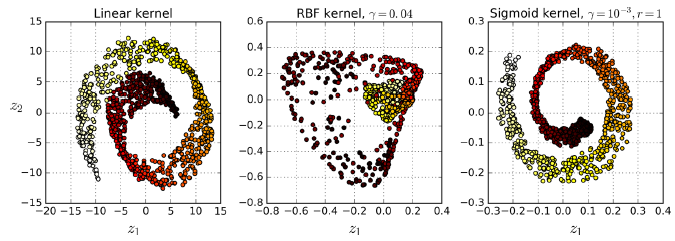
图8-10. 使用不同核将蛋糕卷数据集投影到2维
8.4.1 核的选择与超参数的调整(Selecting a Kernel and Tuning Hyperparameters)
这有两种情况。
第一种情况,尽管kPCA是无监督算法,没有明显的衡量指标帮助我们调整参数。减少降维的下一步往往是监督学习,比如分类。这就可以选择使得分类效果最好的参数。
第二种情况,完全是无监督学习,这是可以选择使得重构误差(reconstruction error)最小的超参数。
8.5 局部线性嵌入(Locally Linear Embedding,LLE)
LLE是另一种强大的非线性降维(nonlinear dimensionality reduction,NLDR)技术。该算法并不像PCA那样依赖投影,而是首先寻找每一个训练两本与其k个近邻(closest neighbors,c.n.)的线性关系,然后找到数据集的一种低维表示,其能够将这些近邻关系最好地保持。该算法对于卷曲数据集表现很好,尤其是噪声不多的情况下。
from sklearn.manifold import LocallyLinearEmbedding lle = LocallyLinearEmbedding(n_components=2, n_neighbors=10) X_reduced = lle.fit_transform(X)
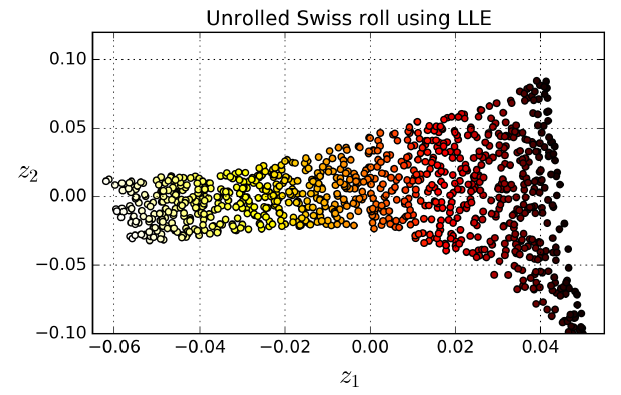
图8-12. 展开蛋糕卷数据集
如图8-12所示,蛋糕卷被展开了。然而,如果数据的范围过大,距离并不能良好的保持下来:蛋糕卷左侧被挤压,右侧被拉伸。
LLE工作方式如下:
首先,对于每一个训练样本$X^{(i)}$,识别出其$k$近邻,然后建立$X^{(i)}$与其$k$近邻的线性方程。
LLE第一步,局部线性关系模型:
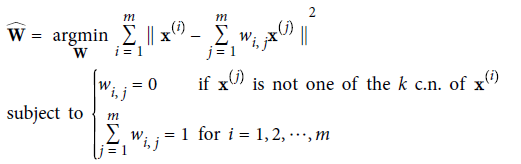
然后,将训练集映射(这里是映射,也就是map,不是前面的投影projecting)到d维空间(d<n),并尽可能地保持局部关系。
LLE第二步,降维并保留局部关系:

8.6 其它的降维技术(Other Dimensionality Reduction Techniques)
- Multidimensional Scaling (MDS),保持样本距离降维。
- Isomap,在样本的近邻间创建图,降维时保持geodesic distances。
- t-Distributed Stochastic Neighbor Embedding (t-SNE),降维时保持相似样本接近,不相似样本远离。这在样本可视化时很有用,尤其是样本簇的可视化。
- Linear Discriminant Analysis (LDA),这实际上是一个分类算法,在训练的时候可以学习到最能区别类别的坐标轴,然后用这些坐标轴定义超平面并将样本映射于其上。其优势是可以尽可能地保持样本点分离,可以再应用分离算法(比如SVM)之前使用LDA。
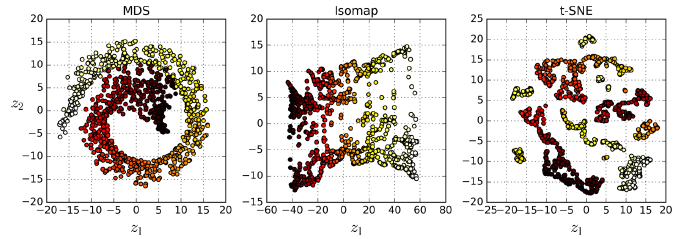
图8-13. 不同算法将蛋卷数据集映射到2维



