过去的10年里,信息技术的发展(包括:大容量电子存储媒介、因特网、分布式数据库技术)和开放性数据共享政策(大范围的数字化工作、数据对公共普通用户的开放性使用)为生物多样性信息的产生、维持、传播和使用带来了革命性的发展(Bisby, 2000; Bisby et al., 2002; Causey et al., 2004; Edwards et al., 2000; Krishtalka and Humphrey, 2000; Krishtalka et al., 2002; Oliver et al., 2000; Smith et al., 2000),产生了生物多样性信息学(Biodiversity Informatics)这一非常活跃的研究领域,并且保持着相当乐观的发展前景(Godfray, 2002; Godfray, 2007; Godfray et al., 2007; Graham et al., 2004)。生物多样性信息学指利用IT技术,对生物有机体基础数据(Primary data)的管理、算法的探究、分析和解释,尤其是在物种水平上的应用(Berendsohn and Geoffroy, 2007; Johnson, 2007; Soberon and Peterson, 2004)。因此,它涉及到对信息的捕获、存储、提交、回溯和分析,它主要关注单个有机体、居群和分类群以及它们的相互关系。它的信息内容覆盖了系统学、进化生物学、居群生物学、行为科学,以及从传粉生物学到寄生病和植物社会学等的大生态等领域。目前生物多样性信息学最重要的研究和活动目标是为生物多样性和全球变化研究提供一个合理的信息管理的下层建筑。
对生物学数据的信息模型和标准的研究在过去几十年是一个非常重要的主题(Berendsohn, 1997; Le Renard, 2000; Pullan et al., 2000; Ytow et al., 2001; Zhong et al., 1996)。IUBS(International Union of Biological Sciences)分类学数据委员会有专门的任务和论坛来讨论相关的信息交换标准。但是,过去的数据标准实践和应用的失败表明,我们对这些问题的复制性缺乏足够的估计,需要更多专门的研究人员和相互地交流来解决它们(Berendsohn and Geoffroy, 2007)。
达尔文核心(Darwin Core:简称DwC)(纪力强 et al., 2005)是生物多样性信息学中目前应用最为广泛,也是非常重要的标准之一。它是一套用于描述生物有机体分布及其相关采集信息的规范。这个规范由一个预先定义的词汇集组成。其中包括了描述生物有机体的学名、分布的地点、标本采集事件(包括采集人、采集时间和地点)等信息。这个标准提出主要是促进不同研究机构和数据库间有效地交换生物有机体分布和相关的采集信息。
然而,由于这一标准所涉及到的生物学信息存在复杂性,并且在形成过程中中又常以不同形式的“修订版”或“扩展版”出现,从而使该标准出现多个版本并存,给我们认识和理解DwC,以及在实践中如何有效地应用带来困难。因此,本文的目是对DwC这个标准的来源、历史变化过程、内涵和实践应用进行较为详细的讨论,以期为从事生物多样性信息学及相关领域的研究人员提供一个参考。
一、 什么是Darwin Core?
DwC的提出是基于科学家和信息技术人员一个非常简单的想法。我们可以想象一下如下的情形。有两个研究组织通过数据库管理着同样一份采集的多个复份标本。这两个组织会在它们各自的数据库中存储非常相似或者几乎完全相同的信息,但是这些信息可能以不同的格式进行存储。以采集人为例:其中一个研究机构的数据库中给这个数据项定义取名为“Gatherer”,而另外一个研究机构的数据库则把同样的数据项处理为“Collector”(极端的情况是某些中文环境运行的数据库把该数据项处理为“Caijiren”)。当这种情况发生在成千上万的研究项目和数据库的时候,任何人都很难去处理这些数据库中的细微差别,我们也不难想象在数据交换和信息共享过程中将会面临的巨大困难。DwC提出的基本目的就是来解决这些相同数据项或信息内容在不同研究机构的数据库中应用不同表达方式的问题。
这些机构自身的数据库可能包括许多不同数据格式和类型的信息,以及为了满足机构管理所需要的特定信息内容。而DwC标准的价值在于它仅仅包括生物学数据库中最“核心”(Core)的信息成分,也就信息元素中共有和相同的内容,来促进这些研究机构间对“核心”生物学采集信息的交换。因为无论是一份动物标本还是一份植物标本,每一个自然历史采集都有它的采集地点、采集时间来反映这一生物体在一定时期的地理分布情况。每个采集都带有标签,可能是手写的,或打印的。它记载着这份采集特有的信息。尽管采集及其它们的标签千差万别,但是几乎所有的生物采集和观察数据在内容上都有共同点,能够通过一些类似或共有的指令在不同的数据集中进行搜集和回溯。DwC试图提供一些标识反映这些共同点,而不是考虑存储这些记录内容的具体形式。对这些标本所共有信息在数据库信息交换过程中表达方式的规范化和统一性就是DwC标准的具体内容。
通过以上的描述,我们对DwC的定义做如下的归纳:Darwin Core是设计用来促进数据库间有效交换生物有机体在自然界的分布信息的数据共享标准。这个标准是通过定义一系列表达特定数据信息的词汇(Concepts/Terms)来实现。它以分类群(Taxon)为最基本的信息单位。有机体的分布信息又体现在各种形式的数据上,例如:观察记录(Observation records),标本(Specimens)以及其它野外调查的样品(Living Collection)等。DwC所定义的这些词汇在一些计算机技术环境下,常常有被称为属性(Properties)、成分(Elements)、字段(Fields)或者类(Class)。在数据库环境下,这些词汇常常是与数据项一一对应。
因此DwC标准形成的基础就是任何自然历史采集和观察数据信息的共同属性,这个标准的目的就是为这些信息的共享和交换机制提供一个指南性原则。它提供的是一个关于数据概念的描述规范,支持针对原始数据的检索和集成,是描述自然历史标本和观察数据特性的最小集合(纪力强 et al., 2005)。
二、DwC的发展历史
DwC最初来源于美国国家自然基金资助的研究项目:生物学采集和分类学数据信息检索协议测试项目(An Experimental Z39.50 Information Retrieval Protocol Test Bed for Biological Collection and Taxonomic Data)。该项目的主要目的是示范在生物标本和权威物种数据信息化过程中采用相关数据标准的实际效果。从而使用户可以通过互联网查询多个标本馆的标本,促进分类学研究和对生物多样性基础数据的科普和研究应用。但是当时还没有使用Darwin Core这个数据项,而是使用的“Z39.50 profile“。后来,由Allen Allison于1998年在美国堪萨斯大学(University of Kansas)举办的ZBIG (Z39.50 Biology Implementers Group)会议上提出。也就是我们所说的DwC原型概念(prototypical Darwin Core)。这个会议上,科学家们讨论了Z39.50在自然历史采集数据中的信息成分问题,以及应用“Darwin Core Profile”和以前存在的都柏林核心元数据(Dublin Core Metadata:简称DC)在概念上和应用上的相似性。因此,DwC是基于都柏林核心元数据标准,它应该被看做是都柏林核心元数据标准在生物多样性信息应用上的扩展。但是它减少了在不同技术实现条件下,比如XML (eXtensible Markup Language),RDF(Resource Description Framework),或者是简单的CSV(Comma Separated Values)文本文件,数据项在语义上的耦合性。
此后,堪萨斯大学自然历史历史博物馆的生物多样性研究小组发起了物种分析者项目(Species Analyst project)。该项目的目标就是开发一些促进世界自然历史采集和观察信息共享的工具和标准。DwC比较完整的体系和概念正是在通过这个项目形成。
DwC正式向外发布的版本是与分布式信息检索协议项目(Distributed Generic Information Retrieval protocol: 简称DiGIR)(目前已被TDWG Access Protocol for Information Retrieval : 简称TAPIR替代)合作形成的,就是现在的DwC1.2。在DiGIR项目中,研究人员把DwC表达为XML模式(XML Schema)的形式,也就是我们后来所称的达尔文核心模式(Darwin Core Schema)。达尔文核心模式向使用者推荐了一个规范,可以用来组织自然历史标本和观察数据库。它也提供了一个从支持达尔文核心的信息服务器中获取数据内容、结构和格式的指南(纪力强 et al., 2005)。
最早正式使用DwC标准的生物多样性信息系统是UC Berkeley的哺乳动物网络信息系统项目(Mammal Networked Information System Project: 简称MaNIS)。MaNIS项目的研究人员在DwC1.2的基础上进行了改善,形成了后来的DwC1.21。在这个版本中已经包括了地理空间和标本管理扩展的部分,形成了后来所说的达尔文核心扩展(Darwin Core Extension)。
DwC的最新版本是DwC1.4。目前由生物多样性信息标准TDWG(Biodiversity Information Standards: TDWG)来维护和管理。该组织是专门致力于生物多样性信息标准的建立、推广和应用。并且有专门的网站和信息系统用于这些标准及其相关电子资源的管理。其中有专门的DwC任务组(Darwin Core Task Group),以及专门的网页(http://www.tdwg.org/standards/450/ )来记录该标准形成的过程,与它相关的电子资源、文件、用户的评论、对标准的修改意见等。DwC无论从开始的1.2版还是到现在的1.4版一直是被TDWG当做一个草案标准(Draft standard)来推行,这意味着专家们认为它尚不完全成熟,还需要在实践过程中不断的修正。可喜的是在经过将近10余年的讨论和不断完善后,DwC被TDWG在2009年10月9号批准为正式标准(表1)。
表1. 达尔文核心的发展历史
Table 1. History of the Darwin Core
| 名称 Name | 命名空间 Namespace | 数据项的数目 Number of terms | XML架构 XML Schema | 发布时间 Date Issued |
| Darwin Core 1.0 | 无 | 24 | (X39.50 GRS-1)) | 1998 |
| Darwin Core 1.2(经典版) | 46 | http://digir.net/schema/conceptual/darwin/2003/1.0/darwin2.xsd | 2001-09-11 | |
| Darwin Core 1.2(应用版) | 63 | http://digir.net/schema/conceptual/darwin/manis/1.21/darwin2.xsd | 2003-03-15 | |
| Darwin Core OBIS | 27 | http://iobis.org/obis/obis.xsd | 2005-07-10 | |
| Darwin Core 1.4(草稿) | 45 | http://rs.tdwg.org/dwc/tdwg_dw_core.xsd | 2005-07-10 | |
| Darwin Core数据项(属性) | 172/181 | http://rs.tdwg.org/dwc/xsd/tdwg_dwcterms.xsd | 2009-10-09 |
表格资料来自:http://en.wikipedia.org/wiki/Darwin_Core#cite_note-history-5
三、DwC的内涵
DwC标准建立的动机是促进生物有机体采集信息的共享和整合。然而在现实中,该标准有比较广泛的含义和应用上的灵活性。它是在一个比较灵活的框架下来提供一个具有强扩展性和应用性的词汇列表,从而提供一个稳定和标准的语义解释,并且最大化这些词汇在不同环境下的重复使用。这也就是我们所说的标准在实践应用中的兼容性问题。DwC标准在实践应用中包括三个主要的版本,分别是简单DwC(Simple Darwin Core)、标准DwC(1.2/1.4)、DwC扩展(Darwin Core Extension)。
简单DwC是DwC的简化版本,它以简化的方式来处理生物类群在自然界的分布信息。如果我们听见大家说“请按DwC标准来格式化你的数据”,那么他一般很可能指的就是简单DwC。简单DwC的两个主要原则是简单性(Simplicity)和灵活性(Flexibility)。简单性表现在它把数据结构都想象为一个二维的平面数据表,二维表的行和列分别对应为数据的属性和属性值。换句话说,通过简单DwC,每一个数据记录(Data Record)可以用一个二维的平面表或电子表格文件(例如Excel, CSV格式)来表示。灵活性表现在它并不是要求每一个字段都是必须的。因此你可以根据自身数据的情况来选择性的使用简单DwC所预先界定的数据项。这些数据项又可以归纳为处理分布、鉴定或者是记录等如下的10类信息。
1、记录水平上的数据项(Record-level Terms)
2、分布数据项(Occurrence Terms)
3、事件数据项(Event Terms)
4、自然地理背景数据项(GeologicalContext Terms)
5、分布地点数据项(Location Terms)
6、鉴定的数据项(Identification Terms)
7、分类群的数据项(Taxon Terms)
8、资源关系的数据项(ResourceRelationship Terms)
9、度量单位或什么的数据项(MeasurementOrFact Terms)
10、数据项类型 (Type Vocabulary Terms)
简单DwC比较适合缺乏在线数据处理系统时多个数据提供者参与,工作周期性长,数据零散的情况。
标准DwC是大部分在线应用系统所广泛采用的,它与以前版本的重要区别在如下几个方面:
1. 标准DwC的基本目的是对所有生物学数据具有更广泛的适用性,主要涵盖的数据是生物学尤其是标本数据;
2. DwC的基础是一系列可以重复使用的概念(Reusable concepts)(或者叫数据项:Terms),而不是一系列的XML模式设计;
3. 这个概念的列表是通过规范的RDF来维护。其它的一些文档用来说明这些词汇的含义和在实际应用,以及在扩展使用时的机制;
4. 这个列表的数据项来自以前DwC XML模式的定义(包括标本馆管理方面,分布信息的GIS表达,古生物学扩展信息等),以及一些推荐用来描述观察数据、动物的移动、栽培植物的野生近缘种,遗传资源和敏感性数据方面。
由于DwC标准本质是关注生物采集信息的共性,所以当面临要处理研究机构特定或某些与采集相关的研究活动,如标本馆的管理,利用地理信息系统对标本分布点的显示,与物种演化相关的古生物学等信息时,我们就需要在DwC标准词汇的基础上添加新的词汇来描述和管理这些信息。这就是我们所说的DwC扩展,它允许标准的实践者们在“核心”信息表达方式“统一”的前提下的灵活性。DwC扩展也经常是由一些比较大的生物多样性信息系统提出,比如:MaNIS (http://manisnet.org/)、HerpNet (http://www.herpnet.org/ )、ORNIS(http://olla.berkeley.edu/ornisnet/ )、FishNet2 (http://www.fishnet2.net/index.html)和OBIS(http://www.iobis.org/ )。目前有不同研究项目和机构实践的扩展成分有:用在标本馆用于管理标本的借阅、登记、出库和入库等信息的标本管理扩展(Curatorial Extension);用于对标本空间地理坐标信息处理的地理空间扩展(Geospatial Extension);用于分类群相应的化石、历史演化记录等信息的古生物学扩(Paleontology Extension)以及描述两个分类群相互关系(互惠、共生等)的相互作用扩展(Interaction Extension)。如果你打算在你研究领域应用DwC扩展,那么你需要首先熟悉DwC命名空间(Namespace)的约定(Darwin Core Namespace Policy)。一般来说,在提出一个新词汇之前,我们需要考虑这个新词汇是否与已经有的那些词汇相互冲突,是否相互兼容。
由于自然历史采集信息本身信息的复杂性,以及在研究、管理和应用上的多样性,科学家们认识到DwC在处理这些信息上有较大的局限性,因此,他们又在DwC的基础上提出了一个包括1,200词汇列表的ABCD(Access to Biological Collections Data)标准。该标准几乎覆盖了采集信息相关的所有可能数据项。因此,与ABCD比较,DwC相对比较简单容易实施,而且大部分DwC中的成分都可以和ABCD中互对应(DwC和ABCD的映射关系见:http://rs.tdwg.org/dwc/terms/history/dwctoabcd/index.htm )。ABCD和DwC都是一种语义数据标准,二者都是为了描述自然历史标本和观察数据。但是二者的使用范围不同。通常来说,DwC被看作是ABCD的子集,是上述数据的的共同点的最小集合的轮廓描述。而ABCD是条目级(Item-level)的数据描述标准。一个条目可能是一份标本、一株培养物、一个活动生物体或是一个生物体的观察。ABCD提供了全面的基础结构,因此它的组成很庞大,并且高度结构化。因此会产生一定的数据冗余。根据ABCD和DwC的不同特点,它们分别被应用到数据交换的不同方面。ABCD作为一个分布式查询返回结果的标准格式,被作为从保藏品数据库返回查询结果的结果模式(Result-schema)。而非等级化、元素数目相对较少的达尔文核心则被用于查询这些数据的过程。本质上说,ABCD是对信息更为细化和全面的考虑,但是在实践实施上有比较大的复杂性和操作上的困难,从而降低了作为一个标准的可操作性(纪力强 et al., 2005)。
由于XML即可扩展标记语言(eXtensible Markup Language)的广泛应用,使异构数据库间信息共享和交换问题得到极大的改善。XML是一种工业标准,它使用文档类型定义(Document Type Definition: DTD)或者模式(Schema)来描述数据。因此,从DiGIR项目开始,DwC就经常使用达尔文核心模式(DwC Schema)的表达标准的定义格式和规范(图1)。它也提供了一个从支持达尔文核心的信息服务器中获取数据内容、结构和格式的指南。
Fig1. Illustration of part XML file for Darwin Core Schema
图1. 简单达尔文核心XML模式片段文件示例
如上所述,DwC本质上是一种语义标准。它本身并不对数据库结构(Database Schema)进行约束。因此,在实践中应用DwC并不意味着在采集数据库设计中应该包括多少个数据表,表中有多少个数据项,以及表间的关联关系是如何的。但是由于DwC模式表达方式的应用,在数据结构和应用程序编程过程中能够在数据库表、数据项、类和属性间相对应的映射关系(图1)。对应不同研究机构和数据库采用DwC标准的意义在于:1、使用相同的方式描述数据;2、整合来自多个数据源的数据;3、具有这些数据分析,做出科学和可靠的决策。
四、DwC的适用范围和特点
DwC的每一个数据项都有一个定义和解释性的说明,目的是让用户在不同应用程序和学科间一致性地应用这些数据项。对这些数据项的讨论、重新界定、应用的例子等内容可以通过DwC网站数据项的评论(Comments)链接获取详细的信息。这种方式表明标准中所定义的数据项和实际操作是有差异的。例如:在数据项的定义中并没有明确指定它们的数据类型(Data types)和数据值的约束条件(Data constraints),但是定义给出了如何设置数值限制条件的建议。
DwC标准所覆盖的核心内容包括:1、任何生物学对象或数据的采集;2、与生物采集数据相关的数据项;3、与其它生物多样性相关数据标准的兼容性;4、便于其它生物学数据成分或属性的增加。为了避免应用中的混淆,DwC也明确了该标准所不适用的范围:1、它并不是一个数据交换的协议;2、它不涉及非生物多样性相关的数据;3、它也不涉及纯粹的分类学数据。
DwC适合的用户群体包括:1、生物多样性数据的拥有者(研究所,大学,研究人员);2、生物多样性数据的使用者;3、采集数据管理系统的开发者;4、其它TDWG兴趣和任务小组的成员;5、生物学采集信息共享协议的开发者(例如: TAPIR);6、生物多样性信息系统的开发者。
TDWG小组通过邮件列表(http://lists.tdwg.org/mailman/listinfo/tdwg)、新闻组和维基百科(http://wiki.tdwg.org/twiki/bin/view/DarwinCore/WebHome )等多种方式让用户广泛参与DwC的制定、修改和讨论。你也可以向TDWG的DwC网站提交XML模式文件来表达你对标准扩展应用的意见。从而是用户不仅仅是一个使用者而且针对实践中不符合需要的标准内容可以发表自己的观点。
五、如何使用DwC
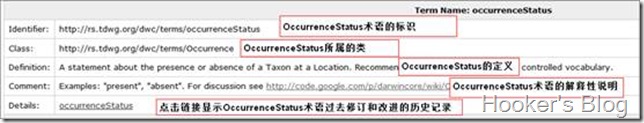
为了有效实施DwC标准,我们需要知道如何使用相关的电子资源和文件。DwC最有价值的在线资源(http://rs.tdwg.org/dwc/terms/index.htm#theterms)由TDWG管理和维护。这个在线资源提供了所有数据项的列表和单个数据项简明扼要的定义、说明、例子及其该数据项形成的历史和修改情况(图2)。
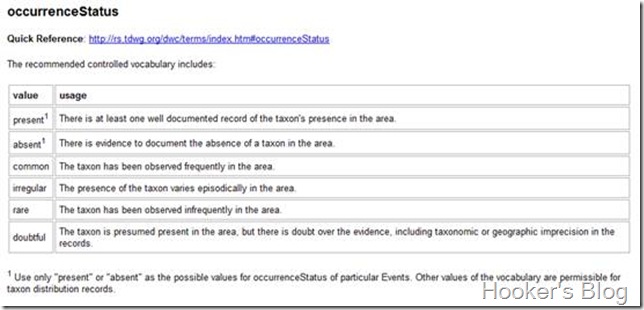
以occurrenceStatus数据项为例,它是用来描述一个分类群在某个地点分布的情况。DwC推荐使用可选值(Controlled vocabulary)的方式来描述。也就是说我们可以使用有(present)、没有(absent)、常见(common)、异常(irregular)、稀少(rare),存疑(doubtful)等方式来描述分布的状态(图3)。
Fig 2. Example of Darwin core terms at online document
图2. Darwin Core数据项在TDWG网络文档上示例和说明
Fig 3. Controlled vocabulary of term occurrenceStatus
图3. occurrenceStatus的可选值及其说明(常被称为controlled vocabulary)
在使用DwC的时候,我们应该注意以下的一些原则:
1. 任何DwC的词汇都可以用作数据库中字段的名称;
2. 每一条记录不允许有重复的字段;
3. 尽量不要使用“类”词汇,比如数据集(DataSet)、地点(Location)、事件(Event)、分类群(Taxon)等,来作为字段名;
4. 尽可能提供较多的字段来细化相关的数据信息;
5. 使用dcterms:type字段来表示数据库信息记录所属的类别;
6. 使用basisOfRecord字段来指明数据记录所属的类型:例如:腊叶标本、化石标本、活体标本、观察数据、分类群名称、命名学行为、分类群名称的用法等;
7. 在使用实践中,尽可能在数据字段中填入一些相应的示例数据,便于用户明确这些字段的含义;
8. 尽量使用DwC标准中对某些数据项推荐使用的可选值;
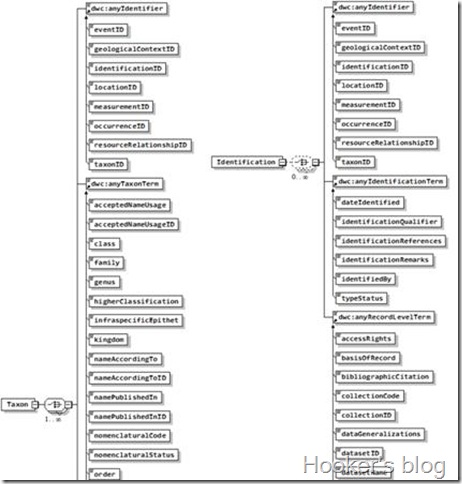
TDWG已经给我们提供了非常方便的DwC模式定义文件(*.xsd),因此我们可以使用相关的XML数据工具(如Altova XMLSpy)从XML定义文件转换为相应的数据库结构(图4,5)。
Fig 4. Example of Taxon and Identification XML Schema Definition(.xsd) Schema View
图4. 分类群和鉴定XML模式定义文件结构示例
Fig 5. Example of Taxon and Identification Database Schema
图5. 分类群和鉴定数据库结构示例
目前国际国内有非常多的生物多样性信息系统都采用了DwC标准。国外的如:全球生物多样性信息网络GBIF (Global Biodiversity Information Facility)、澳大利亚生命百科ALA(Atlas of Living Australia)、海洋生物地理信息系统OBIS (Ocean Biogeographic Information System)、澳大利亚动物采集在线系统OZCAM (Online Zoological Collections of Australian Museums)、哺乳动物信息网络MaNIS (Mammal Networked Information System);国内的有中国数字植物标本馆CVH(Chinese Virtual Herbarium)、中国动物信息网(http://www.animal.net.cn/)等。
六、结束语
信息系统数据共享过程中一般涉及到三类标准:一是数据标准(Data standards),它处理的问题是我们需要共享什么样的数据;二是协议(Protocols),它处理的是这些信息如何进行共享;三是元数据标准(Metadata standards),它向用户提供一个信息摘要,告知用户数据系统可以给他提供什么样的信息。数据标准是首要和最基础的,它是实现协议和元数据标准的前提。
近年来,信息化工作的重视和技术条件的发展促使科学家越来越重视相关标准的实践问题。例如:国内已经成立了专门处理生物学信息化标准的组织—全国生物信息标准化技术委员会(The National Technical Committee for Biological Information Standardization),来帮助相关项目和研究人员处理生物学信息标准及其相关的问题。该委员会是由国家标准化管理委员会领导和管理的从事全国性生物信息标准化工作的技术工作组织,以促进和加强生物信息资源跨国界、跨部门的整合、共享、服务和应用为目的。
因此,为了信息化数据得到更加广泛地使用,并且在不同应用环境和研究领域发挥它最大的价值,数据提供者、管理者和使用者都需要考虑采用一致的语言来描述这些基础信息和数据。
参考文献
Berendsohn, W.G. 1997. A taxonomic information model for botanical databases: the IOPI model. Taxon 46:283-309.
Berendsohn, W.G., & Geoffroy, M. 2007. Networking taxonomic concepts – uniting without “unitary-ism”. Pp. 13-22 in: Curry, G., & Humphries, C., (eds), Biodiversity Databases: From Cottage Industry to Industrial Networks. Systematics Association Special, Taylor & Francis, Boca Raton, FL. p 13-22.
Bisby, F.A. 2000. The Quiet Revolution: Biodiversity Informatics and the Internet. Science 289:2309-2312.
Bisby, F.A., Shimura, J., Ruggiero, M., Edwards, J., & Haeuser, C. 2002. Taxonomy, at the click of a mouse. Nature 418:367-367.
Causey, D., Janzen, D.H., Peterson, A.T., Vieglais, D., Krishtalka, L., Beach, J.H., & Wiley, E.O. 2004. Museum Collections and Taxonomy. Science 305:1106b-1107.
Edwards, J.L., Lane, M.A., & Nielsen, E.S. 2000. Interoperability of Biodiversity Databases: Biodiversity Information on Every Desktop. Science 289:2312-2314.
Godfray, H.C.J. 2002. Challenges for taxonomy. Nature 417:17-19.
Godfray, H.C.J. 2007. Linnaeus in the information age Nature 446:259.
Godfray, H.C.J., Clark, B.R., Kitching, I.J., Mayo, S.J., & Scoble, M.J. 2007. The Web and the Structure of Taxonomy. Systematic Biology 56:943-955.
Graham, C.H., Ferrier, S., Huettman, F., Moritz, C., & Peterson, A.T. 2004. New developments in museum-based informatics and applications in biodiversity analysis. Trends in Ecology & Evolution 19:497-503.
Johnson, N.F. 2007. Biodiversity Informatics. Annual Review of Entomology 52:421-438.
Krishtalka, L., & Humphrey, P.S. 2000. Can natural history museums capture the future? Bioscience 50:611-617.
Krishtalka, L., Peterson, A.T., Vieglais, D.A., Beach, J.H., & Wiley, E.O. 2002. The Green Internet: a tool for conservation science. Pp. 143-164 in: Levitt, J.N., (ed), Conservation in the Internet age: strategic threats and opportunities. Island Press., Washington, DC. p 143-164.
Le Renard, J. 2000. TAXIS, a taxonomic information system for managing large biological collections. In: TDWG 2000: Digitizing Biological Collections. 16th Annual Meeting. Taxonomic Databases Working Group, Frankfurt. p 18.
Oliver, I., A. , Pik, J., Britton, M., Dangerfield, R.K., & Colwell, A.J.B. 2000. Virtual Biodiversity Assessment Systems. BioScience 50:441-450.
Pullan, M.R., Watson, M.F., Kennedy, J.B., Raguenaud, C., & Hyam, R. 2000. The Prometheus Taxonomic Model: a Pratical Approach to Representing Multiple Classifications. Taxon 40:55-75.
Smith, A.T., Boitani, L., Bibby, C., Brackett, D., Corsi, F., da Fonseca, G.A.B., Gascon, C., Dixon, M.G., Hilton-Taylor, C., Mace, G., Mittermeier, R.A., Rabinovich, J., Richardson, B.J., Rylands, A., Stein, B., Stuart, S., Thomsen, J., Wilson, Crispen, Edwards, J.L., Lane, M.A., Nielsen, S., E., & Bisby, F.A. 2000. Databases Tailored for Biodiversity Conservation. Science 290:2073b-2074.
Soberon, J., & Peterson, T. 2004. Biodiversity informatics: managing and applying primary biodiversity data. Philosophical Transactions of the Royal Society B: Biological Sciences 359:689-698.
Ytow, N., Morse, D.R., & Roberts, D.M. 2001. Nomencurator: A nomenclatural history model to handle mutiple taxonomic views. Biological Journal of the Linnean Society 73:81-98.
Zhong, Y., Jung, S., Pramanik, S., & Beaman, J.H. 1996. Data model and comparision query methods for interacting classsifications in taxonomic databases. Taxon 45:223-241.
纪力强, 乔慧捷, & 谢本贵 (eds.). 2005. 全球生物多样性网络(GBIF)介绍:组织、活动、项目和信息服务.
网络资源:
1. TDWG标准Darwin Core简介:http://rs.tdwg.org/dwc/index.htm
2. 维基百科 Darwin Core介绍:http://en.wikipedia.org/wiki/Darwin_Core
3. 美国国家自然基金对An Experimental Z39.50 Information Retrieval Protocol Test Bed for Biological Collection and Taxonomic Data项目的介绍 http://nsf.gov/awardsearch/showAward.do?AwardNumber=9811443
4. 中国国家标准化管理委员会正式批复筹建“ 全国生物信息标准化技术委员会”http://www.cnis.gov.cn/zdly/gxjs/jk/xgjg/200809/t20080918_4320.shtml
5. Dublin Core Metadata Initiative(DCMI)介绍 http://dublincore.org/
6. Darwin Core在Google Code上项目主页:http://code.google.com/p/darwincore/
7. International Union of Biological Sciences:IUBS主页:http://www.iubs.org/newiubs/organisation/index.php
澳大利亚数字植物标本馆(Australia Virtual Hebarium: AVH)HISPID(Herbarium Information Standards and Protocols for Interchange of Data)标准介绍:http://hiscom.chah.org.au/wiki/Herbarium_Information_Standards_and_Protocols_for_Interchange_of_Data






 浙公网安备 33010602011771号
浙公网安备 33010602011771号